Model info
| Transcription factor | Ctcf | ||||||||
| Model | CTCF_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 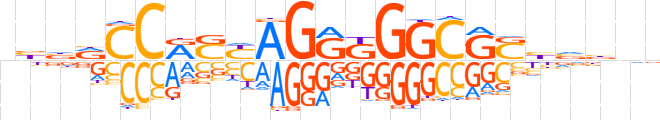 | ||||||||
| LOGO (reverse complement) | 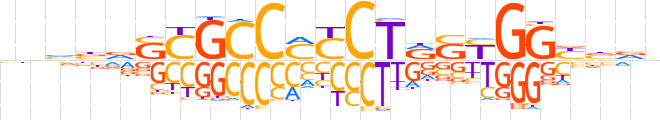 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nbbRCCRSYAGGKGGCGSbvbnb | ||||||||
| Best auROC (human) | 0.999 | ||||||||
| Best auROC (mouse) | 1.0 | ||||||||
| Peak sets in benchmark (human) | 502 | ||||||||
| Peak sets in benchmark (mouse) | 263 | ||||||||
| Aligned words | 495 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | CTCF-like factors {2.3.3.50} | ||||||||
| MGI | MGI:109447 | ||||||||
| EntrezGene | GeneID:13018 (SSTAR profile) | ||||||||
| UniProt ID | CTCF_MOUSE | ||||||||
| UniProt AC | Q61164 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Ctcf expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 10.0 | 29.0 | 14.0 | 45.0 | 5.0 | 46.0 | 19.0 | 61.0 | 21.0 | 62.0 | 17.0 | 72.0 | 3.0 | 46.0 | 13.0 | 32.0 |
| 02 | 3.0 | 5.0 | 26.0 | 5.0 | 39.0 | 41.0 | 65.0 | 38.0 | 12.0 | 16.0 | 22.0 | 13.0 | 17.0 | 28.0 | 148.0 | 17.0 |
| 03 | 13.0 | 5.0 | 48.0 | 5.0 | 31.0 | 5.0 | 47.0 | 7.0 | 44.0 | 30.0 | 151.0 | 36.0 | 17.0 | 2.0 | 47.0 | 7.0 |
| 04 | 5.0 | 93.0 | 6.0 | 1.0 | 2.0 | 34.0 | 2.0 | 4.0 | 14.0 | 252.0 | 15.0 | 12.0 | 0.0 | 54.0 | 0.0 | 1.0 |
| 05 | 0.0 | 20.0 | 0.0 | 1.0 | 4.0 | 429.0 | 0.0 | 0.0 | 1.0 | 22.0 | 0.0 | 0.0 | 0.0 | 18.0 | 0.0 | 0.0 |
| 06 | 0.0 | 2.0 | 3.0 | 0.0 | 317.0 | 9.0 | 134.0 | 29.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 07 | 3.0 | 197.0 | 115.0 | 2.0 | 3.0 | 2.0 | 6.0 | 1.0 | 4.0 | 89.0 | 42.0 | 2.0 | 0.0 | 13.0 | 15.0 | 1.0 |
| 08 | 1.0 | 6.0 | 1.0 | 2.0 | 45.0 | 154.0 | 13.0 | 89.0 | 8.0 | 143.0 | 6.0 | 21.0 | 0.0 | 1.0 | 2.0 | 3.0 |
| 09 | 30.0 | 14.0 | 6.0 | 4.0 | 299.0 | 0.0 | 2.0 | 3.0 | 17.0 | 2.0 | 1.0 | 2.0 | 97.0 | 1.0 | 13.0 | 4.0 |
| 10 | 1.0 | 0.0 | 441.0 | 1.0 | 0.0 | 0.0 | 17.0 | 0.0 | 1.0 | 0.0 | 21.0 | 0.0 | 0.0 | 0.0 | 13.0 | 0.0 |
| 11 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 171.0 | 0.0 | 320.0 | 1.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 12 | 2.0 | 2.0 | 126.0 | 41.0 | 0.0 | 0.0 | 0.0 | 0.0 | 14.0 | 14.0 | 192.0 | 103.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 13 | 0.0 | 0.0 | 15.0 | 1.0 | 0.0 | 0.0 | 16.0 | 0.0 | 2.0 | 1.0 | 313.0 | 3.0 | 0.0 | 0.0 | 144.0 | 0.0 |
| 14 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 12.0 | 10.0 | 438.0 | 28.0 | 0.0 | 0.0 | 4.0 | 0.0 |
| 15 | 0.0 | 11.0 | 0.0 | 1.0 | 2.0 | 8.0 | 1.0 | 0.0 | 29.0 | 398.0 | 0.0 | 16.0 | 0.0 | 29.0 | 0.0 | 0.0 |
| 16 | 2.0 | 0.0 | 28.0 | 1.0 | 125.0 | 2.0 | 318.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 16.0 | 0.0 |
| 17 | 3.0 | 25.0 | 98.0 | 1.0 | 0.0 | 2.0 | 1.0 | 1.0 | 27.0 | 307.0 | 20.0 | 8.0 | 0.0 | 1.0 | 1.0 | 0.0 |
| 18 | 3.0 | 15.0 | 3.0 | 9.0 | 29.0 | 88.0 | 37.0 | 181.0 | 9.0 | 73.0 | 12.0 | 26.0 | 1.0 | 6.0 | 3.0 | 0.0 |
| 19 | 4.0 | 19.0 | 15.0 | 4.0 | 103.0 | 44.0 | 26.0 | 9.0 | 6.0 | 38.0 | 7.0 | 4.0 | 29.0 | 53.0 | 130.0 | 4.0 |
| 20 | 14.0 | 17.0 | 82.0 | 29.0 | 23.0 | 24.0 | 67.0 | 40.0 | 6.0 | 72.0 | 22.0 | 78.0 | 1.0 | 8.0 | 9.0 | 3.0 |
| 21 | 10.0 | 12.0 | 18.0 | 4.0 | 38.0 | 20.0 | 29.0 | 34.0 | 74.0 | 49.0 | 32.0 | 25.0 | 22.0 | 28.0 | 62.0 | 38.0 |
| 22 | 15.0 | 19.0 | 99.0 | 11.0 | 16.0 | 24.0 | 49.0 | 20.0 | 22.0 | 28.0 | 74.0 | 17.0 | 3.0 | 22.0 | 65.0 | 11.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.104 | -0.064 | -0.778 | 0.371 | -1.76 | 0.393 | -0.48 | 0.673 | -0.382 | 0.689 | -0.589 | 0.838 | -2.224 | 0.393 | -0.85 | 0.033 |
| 02 | -2.224 | -1.76 | -0.172 | -1.76 | 0.229 | 0.279 | 0.736 | 0.203 | -0.928 | -0.648 | -0.336 | -0.85 | -0.589 | -0.098 | 1.555 | -0.589 |
| 03 | -0.85 | -1.76 | 0.435 | -1.76 | 0.002 | -1.76 | 0.414 | -1.445 | 0.349 | -0.03 | 1.575 | 0.15 | -0.589 | -2.574 | 0.414 | -1.445 |
| 04 | -1.76 | 1.092 | -1.59 | -3.117 | -2.574 | 0.093 | -2.574 | -1.966 | -0.778 | 2.087 | -0.711 | -0.928 | -4.392 | 0.552 | -4.392 | -3.117 |
| 05 | -4.392 | -0.429 | -4.392 | -3.117 | -1.966 | 2.618 | -4.392 | -4.392 | -3.117 | -0.336 | -4.392 | -4.392 | -4.392 | -0.533 | -4.392 | -4.392 |
| 06 | -4.392 | -2.574 | -2.224 | -4.392 | 2.316 | -1.205 | 1.456 | -0.064 | -4.392 | -4.392 | -4.392 | -4.392 | -4.392 | -3.117 | -4.392 | -4.392 |
| 07 | -2.224 | 1.841 | 1.304 | -2.574 | -2.224 | -2.574 | -1.59 | -3.117 | -1.966 | 1.049 | 0.302 | -2.574 | -4.392 | -0.85 | -0.711 | -3.117 |
| 08 | -3.117 | -1.59 | -3.117 | -2.574 | 0.371 | 1.595 | -0.85 | 1.049 | -1.318 | 1.521 | -1.59 | -0.382 | -4.392 | -3.117 | -2.574 | -2.224 |
| 09 | -0.03 | -0.778 | -1.59 | -1.966 | 2.257 | -4.392 | -2.574 | -2.224 | -0.589 | -2.574 | -3.117 | -2.574 | 1.134 | -3.117 | -0.85 | -1.966 |
| 10 | -3.117 | -4.392 | 2.645 | -3.117 | -4.392 | -4.392 | -0.589 | -4.392 | -3.117 | -4.392 | -0.382 | -4.392 | -4.392 | -4.392 | -0.85 | -4.392 |
| 11 | -4.392 | -4.392 | -2.574 | -4.392 | -4.392 | -4.392 | -4.392 | -4.392 | 1.7 | -4.392 | 2.325 | -3.117 | -4.392 | -4.392 | -3.117 | -4.392 |
| 12 | -2.574 | -2.574 | 1.395 | 0.279 | -4.392 | -4.392 | -4.392 | -4.392 | -0.778 | -0.778 | 1.815 | 1.194 | -4.392 | -4.392 | -3.117 | -4.392 |
| 13 | -4.392 | -4.392 | -0.711 | -3.117 | -4.392 | -4.392 | -0.648 | -4.392 | -2.574 | -3.117 | 2.303 | -2.224 | -4.392 | -4.392 | 1.528 | -4.392 |
| 14 | -4.392 | -3.117 | -3.117 | -4.392 | -4.392 | -4.392 | -4.392 | -3.117 | -0.928 | -1.104 | 2.639 | -0.098 | -4.392 | -4.392 | -1.966 | -4.392 |
| 15 | -4.392 | -1.012 | -4.392 | -3.117 | -2.574 | -1.318 | -3.117 | -4.392 | -0.064 | 2.543 | -4.392 | -0.648 | -4.392 | -0.064 | -4.392 | -4.392 |
| 16 | -2.574 | -4.392 | -0.098 | -3.117 | 1.387 | -2.574 | 2.319 | -3.117 | -4.392 | -3.117 | -4.392 | -4.392 | -4.392 | -3.117 | -0.648 | -4.392 |
| 17 | -2.224 | -0.21 | 1.144 | -3.117 | -4.392 | -2.574 | -3.117 | -3.117 | -0.134 | 2.284 | -0.429 | -1.318 | -4.392 | -3.117 | -3.117 | -4.392 |
| 18 | -2.224 | -0.711 | -2.224 | -1.205 | -0.064 | 1.037 | 0.177 | 1.756 | -1.205 | 0.851 | -0.928 | -0.172 | -3.117 | -1.59 | -2.224 | -4.392 |
| 19 | -1.966 | -0.48 | -0.711 | -1.966 | 1.194 | 0.349 | -0.172 | -1.205 | -1.59 | 0.203 | -1.445 | -1.966 | -0.064 | 0.533 | 1.426 | -1.966 |
| 20 | -0.778 | -0.589 | 0.967 | -0.064 | -0.292 | -0.25 | 0.766 | 0.254 | -1.59 | 0.838 | -0.336 | 0.917 | -3.117 | -1.318 | -1.205 | -2.224 |
| 21 | -1.104 | -0.928 | -0.533 | -1.966 | 0.203 | -0.429 | -0.064 | 0.093 | 0.865 | 0.455 | 0.033 | -0.21 | -0.336 | -0.098 | 0.689 | 0.203 |
| 22 | -0.711 | -0.48 | 1.155 | -1.012 | -0.648 | -0.25 | 0.455 | -0.429 | -0.336 | -0.098 | 0.865 | -0.589 | -2.224 | -0.336 | 0.736 | -1.012 |