Model info
| Transcription factor | Esrrg | ||||||||
| Model | ERR3_MOUSE.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 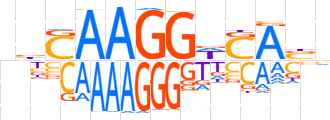 | ||||||||
| LOGO (reverse complement) | 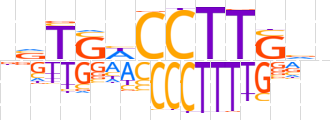 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 12 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nbCAAGGWSAbn | ||||||||
| Best auROC (human) | |||||||||
| Best auROC (mouse) | 0.86 | ||||||||
| Peak sets in benchmark (human) | |||||||||
| Peak sets in benchmark (mouse) | 2 | ||||||||
| Aligned words | 258 | ||||||||
| TF family | Steroid hormone receptors (NR3) {2.1.1} | ||||||||
| TF subfamily | ER-like receptors (NR3A&B) {2.1.1.2} | ||||||||
| MGI | MGI:1347056 | ||||||||
| EntrezGene | GeneID:26381 (SSTAR profile) | ||||||||
| UniProt ID | ERR3_MOUSE | ||||||||
| UniProt AC | P62509 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Esrrg expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 4.0 | 8.0 | 19.0 | 10.0 | 10.0 | 18.0 | 5.0 | 34.0 | 4.0 | 20.0 | 18.0 | 31.0 | 4.0 | 12.0 | 13.0 | 31.0 |
| 02 | 1.0 | 16.0 | 5.0 | 0.0 | 5.0 | 52.0 | 1.0 | 0.0 | 1.0 | 38.0 | 15.0 | 1.0 | 2.0 | 74.0 | 30.0 | 0.0 |
| 03 | 4.0 | 4.0 | 1.0 | 0.0 | 178.0 | 1.0 | 0.0 | 1.0 | 48.0 | 0.0 | 2.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 04 | 225.0 | 3.0 | 1.0 | 2.0 | 5.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 |
| 05 | 7.0 | 0.0 | 225.0 | 2.0 | 1.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 06 | 0.0 | 2.0 | 5.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 3.0 | 222.0 | 3.0 | 0.0 | 2.0 | 1.0 | 0.0 |
| 07 | 0.0 | 0.0 | 0.0 | 2.0 | 5.0 | 0.0 | 0.0 | 2.0 | 41.0 | 10.0 | 41.0 | 136.0 | 3.0 | 0.0 | 0.0 | 1.0 |
| 08 | 2.0 | 34.0 | 12.0 | 1.0 | 0.0 | 7.0 | 2.0 | 1.0 | 1.0 | 38.0 | 0.0 | 2.0 | 0.0 | 105.0 | 27.0 | 9.0 |
| 09 | 3.0 | 0.0 | 0.0 | 0.0 | 160.0 | 7.0 | 14.0 | 3.0 | 34.0 | 2.0 | 4.0 | 1.0 | 12.0 | 1.0 | 0.0 | 0.0 |
| 10 | 16.0 | 110.0 | 53.0 | 30.0 | 0.0 | 7.0 | 1.0 | 2.0 | 2.0 | 12.0 | 3.0 | 1.0 | 0.0 | 1.0 | 0.0 | 3.0 |
| 11 | 11.0 | 0.0 | 7.0 | 0.0 | 33.0 | 52.0 | 23.0 | 22.0 | 19.0 | 17.0 | 14.0 | 7.0 | 6.0 | 6.0 | 8.0 | 16.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.266 | -0.613 | 0.228 | -0.398 | -0.398 | 0.175 | -1.059 | 0.802 | -1.266 | 0.278 | 0.175 | 0.71 | -1.266 | -0.222 | -0.144 | 0.71 |
| 02 | -2.44 | 0.059 | -1.059 | -3.805 | -1.059 | 1.223 | -2.44 | -3.805 | -2.44 | 0.912 | -0.004 | -2.44 | -1.883 | 1.574 | 0.678 | -3.805 |
| 03 | -1.266 | -1.266 | -2.44 | -3.805 | 2.449 | -2.44 | -3.805 | -2.44 | 1.144 | -3.805 | -1.883 | -2.44 | -2.44 | -3.805 | -3.805 | -3.805 |
| 04 | 2.683 | -1.528 | -2.44 | -1.883 | -1.059 | -3.805 | -3.805 | -3.805 | -1.528 | -3.805 | -3.805 | -3.805 | -2.44 | -3.805 | -2.44 | -3.805 |
| 05 | -0.741 | -3.805 | 2.683 | -1.883 | -2.44 | -3.805 | -2.44 | -2.44 | -3.805 | -3.805 | -1.883 | -3.805 | -3.805 | -3.805 | -1.883 | -3.805 |
| 06 | -3.805 | -1.883 | -1.059 | -2.44 | -3.805 | -3.805 | -3.805 | -3.805 | -1.883 | -1.528 | 2.67 | -1.528 | -3.805 | -1.883 | -2.44 | -3.805 |
| 07 | -3.805 | -3.805 | -3.805 | -1.883 | -1.059 | -3.805 | -3.805 | -1.883 | 0.987 | -0.398 | 0.987 | 2.18 | -1.528 | -3.805 | -3.805 | -2.44 |
| 08 | -1.883 | 0.802 | -0.222 | -2.44 | -3.805 | -0.741 | -1.883 | -2.44 | -2.44 | 0.912 | -3.805 | -1.883 | -3.805 | 1.923 | 0.574 | -0.5 |
| 09 | -1.528 | -3.805 | -3.805 | -3.805 | 2.343 | -0.741 | -0.071 | -1.528 | 0.802 | -1.883 | -1.266 | -2.44 | -0.222 | -2.44 | -3.805 | -3.805 |
| 10 | 0.059 | 1.969 | 1.242 | 0.678 | -3.805 | -0.741 | -2.44 | -1.883 | -1.883 | -0.222 | -1.528 | -2.44 | -3.805 | -2.44 | -3.805 | -1.528 |
| 11 | -0.306 | -3.805 | -0.741 | -3.805 | 0.772 | 1.223 | 0.416 | 0.372 | 0.228 | 0.118 | -0.071 | -0.741 | -0.887 | -0.887 | -0.613 | 0.059 |