Model info
| Transcription factor | GRHL2 (GeneCards) | ||||||||
| Model | GRHL2_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 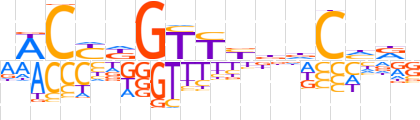 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 15 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | RWCYKGTYYddChRS | ||||||||
| Best auROC (human) | 0.98 | ||||||||
| Best auROC (mouse) | 0.966 | ||||||||
| Peak sets in benchmark (human) | 22 | ||||||||
| Peak sets in benchmark (mouse) | 4 | ||||||||
| Aligned words | 455 | ||||||||
| TF family | Grainyhead-related factors {6.7.1} | ||||||||
| TF subfamily | GRH-like proteins {6.7.1.1} | ||||||||
| HGNC | HGNC:2799 | ||||||||
| EntrezGene | GeneID:79977 (SSTAR profile) | ||||||||
| UniProt ID | GRHL2_HUMAN | ||||||||
| UniProt AC | Q6ISB3 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | GRHL2 expression | ||||||||
| ReMap ChIP-seq dataset list | GRHL2 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 235.0 | 1.0 | 19.0 | 24.0 | 11.0 | 2.0 | 0.0 | 5.0 | 75.0 | 0.0 | 16.0 | 15.0 | 22.0 | 2.0 | 4.0 | 23.0 |
| 02 | 0.0 | 340.0 | 3.0 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 37.0 | 2.0 | 0.0 | 0.0 | 62.0 | 4.0 | 1.0 |
| 03 | 0.0 | 0.0 | 0.0 | 0.0 | 49.0 | 281.0 | 14.0 | 100.0 | 0.0 | 6.0 | 1.0 | 2.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 04 | 6.0 | 0.0 | 43.0 | 0.0 | 87.0 | 2.0 | 67.0 | 131.0 | 6.0 | 0.0 | 8.0 | 1.0 | 2.0 | 1.0 | 85.0 | 15.0 |
| 05 | 0.0 | 0.0 | 101.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 2.0 | 200.0 | 1.0 | 0.0 | 0.0 | 147.0 | 0.0 |
| 06 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 14.0 | 77.0 | 0.0 | 360.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 07 | 3.0 | 4.0 | 0.0 | 7.0 | 1.0 | 36.0 | 0.0 | 42.0 | 0.0 | 0.0 | 1.0 | 0.0 | 11.0 | 86.0 | 1.0 | 262.0 |
| 08 | 2.0 | 2.0 | 0.0 | 11.0 | 11.0 | 32.0 | 11.0 | 72.0 | 0.0 | 1.0 | 1.0 | 0.0 | 20.0 | 79.0 | 38.0 | 174.0 |
| 09 | 0.0 | 2.0 | 29.0 | 2.0 | 17.0 | 22.0 | 13.0 | 62.0 | 5.0 | 2.0 | 37.0 | 6.0 | 38.0 | 29.0 | 99.0 | 91.0 |
| 10 | 36.0 | 1.0 | 12.0 | 11.0 | 20.0 | 5.0 | 1.0 | 29.0 | 73.0 | 5.0 | 78.0 | 22.0 | 37.0 | 10.0 | 47.0 | 67.0 |
| 11 | 1.0 | 162.0 | 2.0 | 1.0 | 0.0 | 21.0 | 0.0 | 0.0 | 0.0 | 124.0 | 4.0 | 10.0 | 1.0 | 125.0 | 3.0 | 0.0 |
| 12 | 0.0 | 0.0 | 0.0 | 2.0 | 127.0 | 188.0 | 6.0 | 111.0 | 0.0 | 4.0 | 1.0 | 4.0 | 2.0 | 1.0 | 8.0 | 0.0 |
| 13 | 55.0 | 1.0 | 70.0 | 3.0 | 123.0 | 2.0 | 23.0 | 45.0 | 9.0 | 0.0 | 6.0 | 0.0 | 15.0 | 7.0 | 84.0 | 11.0 |
| 14 | 1.0 | 60.0 | 138.0 | 3.0 | 3.0 | 2.0 | 1.0 | 4.0 | 2.0 | 39.0 | 140.0 | 2.0 | 2.0 | 6.0 | 50.0 | 1.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 2.102 | -3.035 | -0.395 | -0.165 | -0.927 | -2.491 | -4.32 | -1.676 | 0.964 | -4.32 | -0.563 | -0.626 | -0.251 | -2.491 | -1.881 | -0.207 |
| 02 | -4.32 | 2.471 | -2.14 | -4.32 | -4.32 | -1.676 | -4.32 | -4.32 | -4.32 | 0.262 | -2.491 | -4.32 | -4.32 | 0.774 | -1.881 | -3.035 |
| 03 | -4.32 | -4.32 | -4.32 | -4.32 | 0.541 | 2.281 | -0.693 | 1.25 | -4.32 | -1.505 | -3.035 | -2.491 | -4.32 | -4.32 | -4.32 | -3.035 |
| 04 | -1.505 | -4.32 | 0.411 | -4.32 | 1.111 | -2.491 | 0.851 | 1.519 | -1.505 | -4.32 | -1.233 | -3.035 | -2.491 | -3.035 | 1.088 | -0.626 |
| 05 | -4.32 | -4.32 | 1.26 | -4.32 | -4.32 | -4.32 | -2.14 | -4.32 | -4.32 | -2.491 | 1.941 | -3.035 | -4.32 | -4.32 | 1.634 | -4.32 |
| 06 | -4.32 | -4.32 | -4.32 | -4.32 | -4.32 | -2.491 | -4.32 | -4.32 | -0.693 | 0.99 | -4.32 | 2.528 | -4.32 | -4.32 | -3.035 | -4.32 |
| 07 | -2.14 | -1.881 | -4.32 | -1.36 | -3.035 | 0.235 | -4.32 | 0.388 | -4.32 | -4.32 | -3.035 | -4.32 | -0.927 | 1.1 | -3.035 | 2.211 |
| 08 | -2.491 | -2.491 | -4.32 | -0.927 | -0.927 | 0.119 | -0.927 | 0.923 | -4.32 | -3.035 | -3.035 | -4.32 | -0.344 | 1.015 | 0.289 | 1.802 |
| 09 | -4.32 | -2.491 | 0.022 | -2.491 | -0.503 | -0.251 | -0.765 | 0.774 | -1.676 | -2.491 | 0.262 | -1.505 | 0.289 | 0.022 | 1.24 | 1.156 |
| 10 | 0.235 | -3.035 | -0.843 | -0.927 | -0.344 | -1.676 | -3.035 | 0.022 | 0.937 | -1.676 | 1.003 | -0.251 | 0.262 | -1.019 | 0.499 | 0.851 |
| 11 | -3.035 | 1.731 | -2.491 | -3.035 | -4.32 | -0.296 | -4.32 | -4.32 | -4.32 | 1.464 | -1.881 | -1.019 | -3.035 | 1.472 | -2.14 | -4.32 |
| 12 | -4.32 | -4.32 | -4.32 | -2.491 | 1.488 | 1.88 | -1.505 | 1.354 | -4.32 | -1.881 | -3.035 | -1.881 | -2.491 | -3.035 | -1.233 | -4.32 |
| 13 | 0.655 | -3.035 | 0.895 | -2.14 | 1.456 | -2.491 | -0.207 | 0.456 | -1.12 | -4.32 | -1.505 | -4.32 | -0.626 | -1.36 | 1.076 | -0.927 |
| 14 | -3.035 | 0.742 | 1.571 | -2.14 | -2.14 | -2.491 | -3.035 | -1.881 | -2.491 | 0.314 | 1.585 | -2.491 | -2.491 | -1.505 | 0.561 | -3.035 |