Model info
| Transcription factor | Lhx2 | ||||||||
| Model | LHX2_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 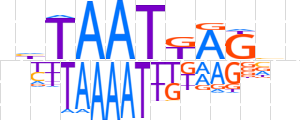 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 11 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nhTAATKAGvn | ||||||||
| Best auROC (human) | 0.804 | ||||||||
| Best auROC (mouse) | 0.934 | ||||||||
| Peak sets in benchmark (human) | 5 | ||||||||
| Peak sets in benchmark (mouse) | 12 | ||||||||
| Aligned words | 531 | ||||||||
| TF family | HD-LIM factors {3.1.5} | ||||||||
| TF subfamily | Lhx-2-like factors {3.1.5.3} | ||||||||
| MGI | MGI:96785 | ||||||||
| EntrezGene | GeneID:16870 (SSTAR profile) | ||||||||
| UniProt ID | LHX2_MOUSE | ||||||||
| UniProt AC | Q9Z0S2 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Lhx2 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 9.0 | 53.0 | 17.0 | 57.0 | 8.0 | 38.0 | 6.0 | 38.0 | 49.0 | 40.0 | 14.0 | 39.0 | 17.0 | 38.0 | 7.0 | 69.0 |
| 02 | 25.0 | 0.0 | 1.0 | 57.0 | 7.0 | 0.0 | 0.0 | 162.0 | 0.0 | 0.0 | 1.0 | 43.0 | 6.0 | 3.0 | 3.0 | 191.0 |
| 03 | 37.0 | 0.0 | 1.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 451.0 | 1.0 | 0.0 | 1.0 |
| 04 | 495.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 05 | 6.0 | 1.0 | 3.0 | 488.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 06 | 1.0 | 0.0 | 5.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 | 2.0 | 0.0 | 3.0 | 16.0 | 209.0 | 261.0 |
| 07 | 2.0 | 0.0 | 2.0 | 0.0 | 16.0 | 0.0 | 1.0 | 0.0 | 201.0 | 0.0 | 16.0 | 0.0 | 175.0 | 2.0 | 82.0 | 2.0 |
| 08 | 19.0 | 5.0 | 319.0 | 51.0 | 0.0 | 0.0 | 2.0 | 0.0 | 6.0 | 10.0 | 80.0 | 5.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 09 | 3.0 | 8.0 | 10.0 | 4.0 | 1.0 | 8.0 | 0.0 | 6.0 | 99.0 | 148.0 | 109.0 | 47.0 | 8.0 | 16.0 | 29.0 | 3.0 |
| 10 | 53.0 | 19.0 | 18.0 | 21.0 | 61.0 | 42.0 | 7.0 | 70.0 | 50.0 | 48.0 | 31.0 | 19.0 | 12.0 | 13.0 | 15.0 | 20.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.213 | 0.525 | -0.597 | 0.597 | -1.326 | 0.195 | -1.598 | 0.195 | 0.447 | 0.246 | -0.786 | 0.221 | -0.597 | 0.195 | -1.452 | 0.787 |
| 02 | -0.218 | -4.398 | -3.124 | 0.597 | -1.452 | -4.398 | -4.398 | 1.638 | -4.398 | -4.398 | -3.124 | 0.318 | -1.598 | -2.232 | -2.232 | 1.802 |
| 03 | 0.169 | -4.398 | -3.124 | -4.398 | -2.232 | -4.398 | -4.398 | -4.398 | -1.768 | -4.398 | -4.398 | -4.398 | 2.66 | -3.124 | -4.398 | -3.124 |
| 04 | 2.753 | -4.398 | -4.398 | -3.124 | -3.124 | -4.398 | -4.398 | -4.398 | -3.124 | -4.398 | -4.398 | -4.398 | -3.124 | -4.398 | -4.398 | -4.398 |
| 05 | -1.598 | -3.124 | -2.232 | 2.739 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -4.398 | -3.124 |
| 06 | -3.124 | -4.398 | -1.768 | -4.398 | -4.398 | -4.398 | -3.124 | -4.398 | -4.398 | -3.124 | -2.582 | -4.398 | -2.232 | -0.656 | 1.892 | 2.114 |
| 07 | -2.582 | -4.398 | -2.582 | -4.398 | -0.656 | -4.398 | -3.124 | -4.398 | 1.853 | -4.398 | -0.656 | -4.398 | 1.715 | -2.582 | 0.959 | -2.582 |
| 08 | -0.488 | -1.768 | 2.314 | 0.487 | -4.398 | -4.398 | -2.582 | -4.398 | -1.598 | -1.112 | 0.934 | -1.768 | -4.398 | -4.398 | -2.582 | -4.398 |
| 09 | -2.232 | -1.326 | -1.112 | -1.973 | -3.124 | -1.326 | -4.398 | -1.598 | 1.147 | 1.547 | 1.243 | 0.406 | -1.326 | -0.656 | -0.072 | -2.232 |
| 10 | 0.525 | -0.488 | -0.541 | -0.39 | 0.665 | 0.294 | -1.452 | 0.802 | 0.467 | 0.427 | -0.006 | -0.488 | -0.936 | -0.858 | -0.719 | -0.437 |