Model info
| Transcription factor | Rest | ||||||||
| Model | REST_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 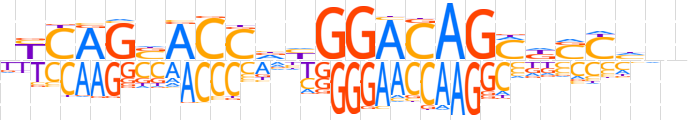 | ||||||||
| LOGO (reverse complement) | 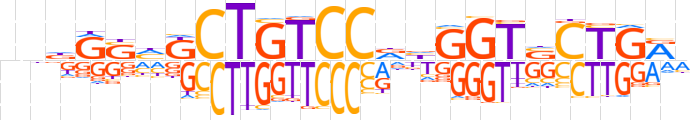 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 24 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nYCAGSACCdhGGACAGMdSYhnv | ||||||||
| Best auROC (human) | 0.999 | ||||||||
| Best auROC (mouse) | 0.998 | ||||||||
| Peak sets in benchmark (human) | 147 | ||||||||
| Peak sets in benchmark (mouse) | 33 | ||||||||
| Aligned words | 332 | ||||||||
| TF family | Factors with multiple dispersed zinc fingers {2.3.4} | ||||||||
| TF subfamily | unclassified {2.3.4.0} | ||||||||
| MGI | MGI:104897 | ||||||||
| EntrezGene | GeneID:19712 (SSTAR profile) | ||||||||
| UniProt ID | REST_MOUSE | ||||||||
| UniProt AC | Q8VIG1 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Rest expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.0 | 12.0 | 8.0 | 24.0 | 9.0 | 18.0 | 3.0 | 14.0 | 0.0 | 15.0 | 14.0 | 47.0 | 3.0 | 15.0 | 3.0 | 147.0 |
| 02 | 0.0 | 9.0 | 2.0 | 1.0 | 7.0 | 38.0 | 7.0 | 8.0 | 0.0 | 20.0 | 0.0 | 8.0 | 2.0 | 216.0 | 4.0 | 10.0 |
| 03 | 2.0 | 0.0 | 6.0 | 1.0 | 269.0 | 6.0 | 6.0 | 2.0 | 4.0 | 4.0 | 5.0 | 0.0 | 9.0 | 4.0 | 11.0 | 3.0 |
| 04 | 4.0 | 5.0 | 273.0 | 2.0 | 5.0 | 2.0 | 4.0 | 3.0 | 2.0 | 7.0 | 17.0 | 2.0 | 2.0 | 3.0 | 1.0 | 0.0 |
| 05 | 3.0 | 0.0 | 10.0 | 0.0 | 3.0 | 8.0 | 2.0 | 4.0 | 20.0 | 186.0 | 59.0 | 30.0 | 2.0 | 2.0 | 3.0 | 0.0 |
| 06 | 17.0 | 2.0 | 7.0 | 2.0 | 189.0 | 1.0 | 3.0 | 3.0 | 55.0 | 12.0 | 6.0 | 1.0 | 31.0 | 1.0 | 2.0 | 0.0 |
| 07 | 1.0 | 289.0 | 1.0 | 1.0 | 4.0 | 3.0 | 7.0 | 2.0 | 2.0 | 8.0 | 4.0 | 4.0 | 0.0 | 5.0 | 1.0 | 0.0 |
| 08 | 1.0 | 3.0 | 1.0 | 2.0 | 6.0 | 273.0 | 12.0 | 14.0 | 1.0 | 5.0 | 4.0 | 3.0 | 0.0 | 1.0 | 6.0 | 0.0 |
| 09 | 2.0 | 1.0 | 4.0 | 1.0 | 167.0 | 30.0 | 46.0 | 39.0 | 7.0 | 8.0 | 7.0 | 1.0 | 1.0 | 3.0 | 9.0 | 6.0 |
| 10 | 29.0 | 59.0 | 7.0 | 82.0 | 3.0 | 3.0 | 2.0 | 34.0 | 8.0 | 35.0 | 2.0 | 21.0 | 0.0 | 18.0 | 4.0 | 25.0 |
| 11 | 0.0 | 0.0 | 40.0 | 0.0 | 3.0 | 1.0 | 110.0 | 1.0 | 0.0 | 0.0 | 14.0 | 1.0 | 1.0 | 2.0 | 159.0 | 0.0 |
| 12 | 0.0 | 0.0 | 4.0 | 0.0 | 1.0 | 0.0 | 1.0 | 1.0 | 3.0 | 1.0 | 317.0 | 2.0 | 0.0 | 0.0 | 2.0 | 0.0 |
| 13 | 3.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 295.0 | 21.0 | 2.0 | 6.0 | 1.0 | 0.0 | 1.0 | 1.0 |
| 14 | 1.0 | 262.0 | 26.0 | 10.0 | 0.0 | 21.0 | 1.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 6.0 | 2.0 | 0.0 |
| 15 | 1.0 | 0.0 | 0.0 | 0.0 | 290.0 | 1.0 | 0.0 | 1.0 | 26.0 | 0.0 | 1.0 | 2.0 | 10.0 | 0.0 | 0.0 | 0.0 |
| 16 | 3.0 | 4.0 | 318.0 | 2.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 2.0 |
| 17 | 0.0 | 2.0 | 1.0 | 0.0 | 1.0 | 1.0 | 1.0 | 2.0 | 35.0 | 248.0 | 11.0 | 26.0 | 1.0 | 1.0 | 2.0 | 0.0 |
| 18 | 1.0 | 1.0 | 30.0 | 5.0 | 59.0 | 3.0 | 91.0 | 99.0 | 1.0 | 0.0 | 11.0 | 3.0 | 8.0 | 2.0 | 9.0 | 9.0 |
| 19 | 14.0 | 35.0 | 15.0 | 5.0 | 0.0 | 4.0 | 1.0 | 1.0 | 21.0 | 87.0 | 27.0 | 6.0 | 7.0 | 105.0 | 4.0 | 0.0 |
| 20 | 1.0 | 35.0 | 2.0 | 4.0 | 18.0 | 182.0 | 2.0 | 29.0 | 11.0 | 32.0 | 2.0 | 2.0 | 0.0 | 10.0 | 1.0 | 1.0 |
| 21 | 8.0 | 13.0 | 4.0 | 5.0 | 70.0 | 134.0 | 21.0 | 34.0 | 1.0 | 1.0 | 2.0 | 3.0 | 5.0 | 18.0 | 8.0 | 5.0 |
| 22 | 16.0 | 28.0 | 33.0 | 7.0 | 62.0 | 39.0 | 45.0 | 20.0 | 8.0 | 12.0 | 8.0 | 7.0 | 9.0 | 12.0 | 16.0 | 10.0 |
| 23 | 16.0 | 20.0 | 48.0 | 11.0 | 29.0 | 19.0 | 30.0 | 13.0 | 25.0 | 39.0 | 29.0 | 9.0 | 8.0 | 11.0 | 19.0 | 6.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -4.064 | -0.535 | -0.926 | 0.143 | -0.813 | -0.14 | -1.837 | -0.385 | -4.064 | -0.318 | -0.385 | 0.808 | -1.837 | -0.318 | -1.837 | 1.943 |
| 02 | -4.064 | -0.813 | -2.19 | -2.74 | -1.053 | 0.597 | -1.053 | -0.926 | -4.064 | -0.036 | -4.064 | -0.926 | -2.19 | 2.327 | -1.577 | -0.712 |
| 03 | -2.19 | -4.064 | -1.199 | -2.74 | 2.546 | -1.199 | -1.199 | -2.19 | -1.577 | -1.577 | -1.37 | -4.064 | -0.813 | -1.577 | -0.62 | -1.837 |
| 04 | -1.577 | -1.37 | 2.561 | -2.19 | -1.37 | -2.19 | -1.577 | -1.837 | -2.19 | -1.053 | -0.196 | -2.19 | -2.19 | -1.837 | -2.74 | -4.064 |
| 05 | -1.837 | -4.064 | -0.712 | -4.064 | -1.837 | -0.926 | -2.19 | -1.577 | -0.036 | 2.178 | 1.034 | 0.363 | -2.19 | -2.19 | -1.837 | -4.064 |
| 06 | -0.196 | -2.19 | -1.053 | -2.19 | 2.194 | -2.74 | -1.837 | -1.837 | 0.964 | -0.535 | -1.199 | -2.74 | 0.396 | -2.74 | -2.19 | -4.064 |
| 07 | -2.74 | 2.618 | -2.74 | -2.74 | -1.577 | -1.837 | -1.053 | -2.19 | -2.19 | -0.926 | -1.577 | -1.577 | -4.064 | -1.37 | -2.74 | -4.064 |
| 08 | -2.74 | -1.837 | -2.74 | -2.19 | -1.199 | 2.561 | -0.535 | -0.385 | -2.74 | -1.37 | -1.577 | -1.837 | -4.064 | -2.74 | -1.199 | -4.064 |
| 09 | -2.19 | -2.74 | -1.577 | -2.74 | 2.07 | 0.363 | 0.787 | 0.623 | -1.053 | -0.926 | -1.053 | -2.74 | -2.74 | -1.837 | -0.813 | -1.199 |
| 10 | 0.33 | 1.034 | -1.053 | 1.361 | -1.837 | -1.837 | -2.19 | 0.487 | -0.926 | 0.516 | -2.19 | 0.012 | -4.064 | -0.14 | -1.577 | 0.183 |
| 11 | -4.064 | -4.064 | 0.648 | -4.064 | -1.837 | -2.74 | 1.654 | -2.74 | -4.064 | -4.064 | -0.385 | -2.74 | -2.74 | -2.19 | 2.021 | -4.064 |
| 12 | -4.064 | -4.064 | -1.577 | -4.064 | -2.74 | -4.064 | -2.74 | -2.74 | -1.837 | -2.74 | 2.71 | -2.19 | -4.064 | -4.064 | -2.19 | -4.064 |
| 13 | -1.837 | -2.74 | -4.064 | -4.064 | -4.064 | -4.064 | -4.064 | -2.74 | 2.638 | 0.012 | -2.19 | -1.199 | -2.74 | -4.064 | -2.74 | -2.74 |
| 14 | -2.74 | 2.52 | 0.222 | -0.712 | -4.064 | 0.012 | -2.74 | -4.064 | -4.064 | -1.837 | -4.064 | -4.064 | -4.064 | -1.199 | -2.19 | -4.064 |
| 15 | -2.74 | -4.064 | -4.064 | -4.064 | 2.621 | -2.74 | -4.064 | -2.74 | 0.222 | -4.064 | -2.74 | -2.19 | -0.712 | -4.064 | -4.064 | -4.064 |
| 16 | -1.837 | -1.577 | 2.713 | -2.19 | -4.064 | -2.74 | -4.064 | -4.064 | -4.064 | -4.064 | -2.74 | -4.064 | -4.064 | -4.064 | -2.74 | -2.19 |
| 17 | -4.064 | -2.19 | -2.74 | -4.064 | -2.74 | -2.74 | -2.74 | -2.19 | 0.516 | 2.465 | -0.62 | 0.222 | -2.74 | -2.74 | -2.19 | -4.064 |
| 18 | -2.74 | -2.74 | 0.363 | -1.37 | 1.034 | -1.837 | 1.465 | 1.549 | -2.74 | -4.064 | -0.62 | -1.837 | -0.926 | -2.19 | -0.813 | -0.813 |
| 19 | -0.385 | 0.516 | -0.318 | -1.37 | -4.064 | -1.577 | -2.74 | -2.74 | 0.012 | 1.42 | 0.259 | -1.199 | -1.053 | 1.608 | -1.577 | -4.064 |
| 20 | -2.74 | 0.516 | -2.19 | -1.577 | -0.14 | 2.156 | -2.19 | 0.33 | -0.62 | 0.427 | -2.19 | -2.19 | -4.064 | -0.712 | -2.74 | -2.74 |
| 21 | -0.926 | -0.457 | -1.577 | -1.37 | 1.204 | 1.851 | 0.012 | 0.487 | -2.74 | -2.74 | -2.19 | -1.837 | -1.37 | -0.14 | -0.926 | -1.37 |
| 22 | -0.255 | 0.295 | 0.458 | -1.053 | 1.083 | 0.623 | 0.765 | -0.036 | -0.926 | -0.535 | -0.926 | -1.053 | -0.813 | -0.535 | -0.255 | -0.712 |
| 23 | -0.255 | -0.036 | 0.829 | -0.62 | 0.33 | -0.087 | 0.363 | -0.457 | 0.183 | 0.623 | 0.33 | -0.813 | -0.926 | -0.62 | -0.087 | -1.199 |