Model info
| Transcription factor | Rfx3 | ||||||||
| Model | RFX3_MOUSE.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO |  | ||||||||
| LOGO (reverse complement) | 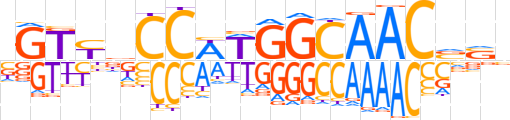 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 18 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | bSbGTTGCCWWGGnRACv | ||||||||
| Best auROC (human) | |||||||||
| Best auROC (mouse) | 0.994 | ||||||||
| Peak sets in benchmark (human) | |||||||||
| Peak sets in benchmark (mouse) | 2 | ||||||||
| Aligned words | 354 | ||||||||
| TF family | RFX-related factors {3.3.3} | ||||||||
| TF subfamily | RFX3 {3.3.3.0.3} | ||||||||
| MGI | MGI:106582 | ||||||||
| EntrezGene | GeneID:19726 (SSTAR profile) | ||||||||
| UniProt ID | RFX3_MOUSE | ||||||||
| UniProt AC | P48381 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Rfx3 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 2.0 | 30.0 | 9.0 | 5.0 | 5.0 | 47.0 | 23.0 | 25.0 | 7.0 | 92.0 | 26.0 | 15.0 | 0.0 | 35.0 | 12.0 | 17.0 |
| 02 | 1.0 | 5.0 | 7.0 | 1.0 | 13.0 | 47.0 | 94.0 | 50.0 | 3.0 | 31.0 | 32.0 | 4.0 | 0.0 | 12.0 | 39.0 | 11.0 |
| 03 | 0.0 | 0.0 | 17.0 | 0.0 | 0.0 | 0.0 | 95.0 | 0.0 | 0.0 | 0.0 | 171.0 | 1.0 | 0.0 | 0.0 | 66.0 | 0.0 |
| 04 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 12.0 | 0.0 | 337.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 05 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 11.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 22.0 | 0.0 | 313.0 |
| 06 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 21.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 20.0 | 4.0 | 287.0 | 13.0 |
| 07 | 0.0 | 20.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 276.0 | 1.0 | 35.0 | 0.0 | 14.0 | 0.0 | 0.0 |
| 08 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 259.0 | 1.0 | 54.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 33.0 | 0.0 | 2.0 |
| 09 | 0.0 | 0.0 | 0.0 | 0.0 | 224.0 | 13.0 | 17.0 | 39.0 | 0.0 | 0.0 | 1.0 | 0.0 | 49.0 | 1.0 | 4.0 | 2.0 |
| 10 | 38.0 | 7.0 | 30.0 | 198.0 | 0.0 | 1.0 | 4.0 | 9.0 | 2.0 | 0.0 | 5.0 | 15.0 | 9.0 | 0.0 | 7.0 | 25.0 |
| 11 | 0.0 | 0.0 | 49.0 | 0.0 | 0.0 | 0.0 | 8.0 | 0.0 | 0.0 | 0.0 | 46.0 | 0.0 | 18.0 | 0.0 | 229.0 | 0.0 |
| 12 | 0.0 | 0.0 | 18.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 26.0 | 3.0 | 303.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 13 | 0.0 | 21.0 | 5.0 | 0.0 | 2.0 | 1.0 | 0.0 | 0.0 | 86.0 | 118.0 | 56.0 | 61.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 14 | 35.0 | 3.0 | 50.0 | 0.0 | 79.0 | 3.0 | 55.0 | 3.0 | 40.0 | 2.0 | 18.0 | 1.0 | 17.0 | 4.0 | 33.0 | 7.0 |
| 15 | 136.0 | 12.0 | 22.0 | 1.0 | 9.0 | 1.0 | 2.0 | 0.0 | 139.0 | 16.0 | 0.0 | 1.0 | 7.0 | 1.0 | 1.0 | 2.0 |
| 16 | 1.0 | 262.0 | 3.0 | 25.0 | 2.0 | 15.0 | 5.0 | 8.0 | 2.0 | 18.0 | 2.0 | 3.0 | 0.0 | 3.0 | 0.0 | 1.0 |
| 17 | 1.0 | 2.0 | 2.0 | 0.0 | 59.0 | 95.0 | 111.0 | 33.0 | 2.0 | 3.0 | 4.0 | 1.0 | 6.0 | 4.0 | 23.0 | 4.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -2.241 | 0.311 | -0.865 | -1.422 | -1.422 | 0.756 | 0.049 | 0.131 | -1.105 | 1.424 | 0.17 | -0.37 | -4.107 | 0.464 | -0.587 | -0.247 |
| 02 | -2.79 | -1.422 | -1.105 | -2.79 | -0.509 | 0.756 | 1.445 | 0.817 | -1.888 | 0.344 | 0.375 | -1.628 | -4.107 | -0.587 | 0.571 | -0.671 |
| 03 | -4.107 | -4.107 | -0.247 | -4.107 | -4.107 | -4.107 | 1.456 | -4.107 | -4.107 | -4.107 | 2.042 | -2.79 | -4.107 | -4.107 | 1.093 | -4.107 |
| 04 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 | -0.587 | -4.107 | 2.719 | -4.107 | -4.107 | -4.107 | -2.79 |
| 05 | -4.107 | -4.107 | -4.107 | -4.107 | -2.79 | -4.107 | -4.107 | -0.671 | -4.107 | -4.107 | -4.107 | -4.107 | -1.888 | 0.006 | -4.107 | 2.645 |
| 06 | -4.107 | -4.107 | -1.628 | -4.107 | -4.107 | -4.107 | -0.04 | -2.79 | -4.107 | -4.107 | -4.107 | -4.107 | -0.088 | -1.628 | 2.559 | -0.509 |
| 07 | -4.107 | -0.088 | -4.107 | -4.107 | -4.107 | -1.628 | -4.107 | -4.107 | -4.107 | 2.52 | -2.79 | 0.464 | -4.107 | -0.437 | -4.107 | -4.107 |
| 08 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 | 2.456 | -2.79 | 0.894 | -4.107 | -2.79 | -4.107 | -4.107 | -4.107 | 0.406 | -4.107 | -2.241 |
| 09 | -4.107 | -4.107 | -4.107 | -4.107 | 2.311 | -0.509 | -0.247 | 0.571 | -4.107 | -4.107 | -2.79 | -4.107 | 0.797 | -2.79 | -1.628 | -2.241 |
| 10 | 0.545 | -1.105 | 0.311 | 2.188 | -4.107 | -2.79 | -1.628 | -0.865 | -2.241 | -4.107 | -1.422 | -0.37 | -0.865 | -4.107 | -1.105 | 0.131 |
| 11 | -4.107 | -4.107 | 0.797 | -4.107 | -4.107 | -4.107 | -0.978 | -4.107 | -4.107 | -4.107 | 0.735 | -4.107 | -0.191 | -4.107 | 2.333 | -4.107 |
| 12 | -4.107 | -4.107 | -0.191 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 | 0.17 | -1.888 | 2.613 | -4.107 | -4.107 | -4.107 | -4.107 | -4.107 |
| 13 | -4.107 | -0.04 | -1.422 | -4.107 | -2.241 | -2.79 | -4.107 | -4.107 | 1.357 | 1.672 | 0.93 | 1.015 | -4.107 | -4.107 | -4.107 | -4.107 |
| 14 | 0.464 | -1.888 | 0.817 | -4.107 | 1.272 | -1.888 | 0.912 | -1.888 | 0.596 | -2.241 | -0.191 | -2.79 | -0.247 | -1.628 | 0.406 | -1.105 |
| 15 | 1.813 | -0.587 | 0.006 | -2.79 | -0.865 | -2.79 | -2.241 | -4.107 | 1.835 | -0.307 | -4.107 | -2.79 | -1.105 | -2.79 | -2.79 | -2.241 |
| 16 | -2.79 | 2.468 | -1.888 | 0.131 | -2.241 | -0.37 | -1.422 | -0.978 | -2.241 | -0.191 | -2.241 | -1.888 | -4.107 | -1.888 | -4.107 | -2.79 |
| 17 | -2.79 | -2.241 | -2.241 | -4.107 | 0.982 | 1.456 | 1.611 | 0.406 | -2.241 | -1.888 | -1.628 | -2.79 | -1.251 | -1.628 | 0.049 | -1.628 |