Model info
| Transcription factor | Rbpj | ||||||||
| Model | SUH_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 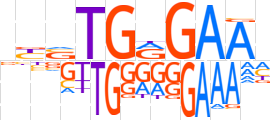 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 10 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | bbSTGRGAAh | ||||||||
| Best auROC (human) | 0.952 | ||||||||
| Best auROC (mouse) | 0.958 | ||||||||
| Peak sets in benchmark (human) | 25 | ||||||||
| Peak sets in benchmark (mouse) | 6 | ||||||||
| Aligned words | 510 | ||||||||
| TF family | CSL-related factors {6.1.4} | ||||||||
| TF subfamily | M {6.1.4.1} | ||||||||
| MGI | MGI:96522 | ||||||||
| EntrezGene | GeneID:19664 (SSTAR profile) | ||||||||
| UniProt ID | SUH_MOUSE | ||||||||
| UniProt AC | P31266 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Rbpj expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 5.0 | 31.0 | 13.0 | 17.0 | 5.0 | 82.0 | 9.0 | 55.0 | 8.0 | 87.0 | 17.0 | 69.0 | 3.0 | 46.0 | 28.0 | 25.0 |
| 02 | 9.0 | 5.0 | 7.0 | 0.0 | 54.0 | 77.0 | 106.0 | 9.0 | 10.0 | 21.0 | 36.0 | 0.0 | 13.0 | 25.0 | 124.0 | 4.0 |
| 03 | 3.0 | 0.0 | 2.0 | 81.0 | 2.0 | 1.0 | 3.0 | 122.0 | 0.0 | 1.0 | 0.0 | 272.0 | 1.0 | 0.0 | 1.0 | 11.0 |
| 04 | 0.0 | 1.0 | 4.0 | 1.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 1.0 | 5.0 | 0.0 | 4.0 | 2.0 | 480.0 | 0.0 |
| 05 | 0.0 | 0.0 | 2.0 | 2.0 | 0.0 | 0.0 | 6.0 | 0.0 | 166.0 | 1.0 | 266.0 | 56.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 06 | 0.0 | 1.0 | 164.0 | 1.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 272.0 | 3.0 | 0.0 | 0.0 | 58.0 | 0.0 |
| 07 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 486.0 | 1.0 | 7.0 | 1.0 | 3.0 | 1.0 | 0.0 | 0.0 |
| 08 | 434.0 | 15.0 | 39.0 | 1.0 | 1.0 | 0.0 | 1.0 | 0.0 | 6.0 | 1.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 09 | 152.0 | 148.0 | 63.0 | 79.0 | 4.0 | 6.0 | 3.0 | 3.0 | 13.0 | 19.0 | 3.0 | 6.0 | 1.0 | 0.0 | 0.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.77 | -0.008 | -0.86 | -0.599 | -1.77 | 0.957 | -1.215 | 0.56 | -1.328 | 1.016 | -0.599 | 0.785 | -2.234 | 0.383 | -0.108 | -0.22 |
| 02 | -1.215 | -1.77 | -1.454 | -4.4 | 0.542 | 0.894 | 1.213 | -1.215 | -1.114 | -0.392 | 0.14 | -4.4 | -0.86 | -0.22 | 1.369 | -1.975 |
| 03 | -2.234 | -4.4 | -2.584 | 0.945 | -2.584 | -3.126 | -2.234 | 1.353 | -4.4 | -3.126 | -4.4 | 2.153 | -3.126 | -4.4 | -3.126 | -1.022 |
| 04 | -4.4 | -3.126 | -1.975 | -3.126 | -4.4 | -2.584 | -4.4 | -4.4 | -4.4 | -3.126 | -1.77 | -4.4 | -1.975 | -2.584 | 2.72 | -4.4 |
| 05 | -4.4 | -4.4 | -2.584 | -2.584 | -4.4 | -4.4 | -1.6 | -4.4 | 1.66 | -3.126 | 2.131 | 0.578 | -4.4 | -4.4 | -3.126 | -4.4 |
| 06 | -4.4 | -3.126 | 1.648 | -3.126 | -4.4 | -4.4 | -3.126 | -4.4 | -4.4 | -4.4 | 2.153 | -2.234 | -4.4 | -4.4 | 0.613 | -4.4 |
| 07 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -3.126 | -4.4 | 2.733 | -3.126 | -1.454 | -3.126 | -2.234 | -3.126 | -4.4 | -4.4 |
| 08 | 2.62 | -0.721 | 0.219 | -3.126 | -3.126 | -4.4 | -3.126 | -4.4 | -1.6 | -3.126 | -3.126 | -4.4 | -3.126 | -4.4 | -4.4 | -4.4 |
| 09 | 1.572 | 1.545 | 0.695 | 0.92 | -1.975 | -1.6 | -2.234 | -2.234 | -0.86 | -0.49 | -2.234 | -1.6 | -3.126 | -4.4 | -4.4 | -4.4 |