Model info
| Transcription factor | ZNF490 (GeneCards) | ||||||||
| Model | ZN490_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO |  | ||||||||
| LOGO (reverse complement) | 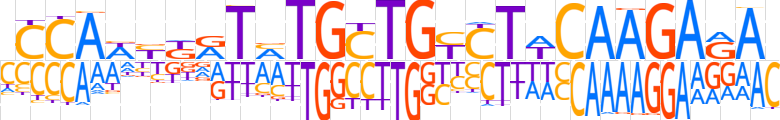 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | GTYTCTTGWAGRCAGCAKAYMRWTGGR | ||||||||
| Best auROC (human) | 0.996 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 421 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF763-like factors {2.3.3.33} | ||||||||
| HGNC | HGNC:23705 | ||||||||
| EntrezGene | GeneID:57474 (SSTAR profile) | ||||||||
| UniProt ID | ZN490_HUMAN | ||||||||
| UniProt AC | Q9ULM2 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF490 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF490 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.0 | 1.0 | 1.0 | 20.0 | 0.0 | 0.0 | 0.0 | 3.0 | 1.0 | 4.0 | 6.0 | 380.0 | 0.0 | 0.0 | 2.0 | 2.0 |
| 02 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 3.0 | 0.0 | 2.0 | 0.0 | 6.0 | 0.0 | 3.0 | 6.0 | 254.0 | 2.0 | 143.0 |
| 03 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 | 1.0 | 0.0 | 262.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 148.0 |
| 04 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 413.0 | 3.0 | 0.0 |
| 05 | 0.0 | 0.0 | 0.0 | 2.0 | 4.0 | 14.0 | 0.0 | 397.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 06 | 0.0 | 0.0 | 1.0 | 3.0 | 0.0 | 0.0 | 0.0 | 14.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 1.0 | 6.0 | 393.0 |
| 07 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 | 2.0 | 2.0 | 405.0 | 1.0 |
| 08 | 1.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 2.0 | 211.0 | 7.0 | 6.0 | 191.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 09 | 204.0 | 1.0 | 4.0 | 3.0 | 7.0 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 | 0.0 | 0.0 | 192.0 | 1.0 | 0.0 | 2.0 |
| 10 | 41.0 | 4.0 | 348.0 | 16.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 3.0 | 1.0 | 1.0 | 0.0 | 4.0 | 0.0 |
| 11 | 15.0 | 0.0 | 27.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 181.0 | 0.0 | 171.0 | 4.0 | 15.0 | 0.0 | 3.0 | 0.0 |
| 12 | 0.0 | 215.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 201.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 |
| 13 | 0.0 | 0.0 | 0.0 | 0.0 | 419.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 14 | 94.0 | 0.0 | 325.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 15 | 0.0 | 95.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 322.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 16 | 2.0 | 0.0 | 0.0 | 0.0 | 417.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 17 | 21.0 | 31.0 | 117.0 | 251.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 18 | 20.0 | 0.0 | 1.0 | 0.0 | 28.0 | 0.0 | 3.0 | 0.0 | 113.0 | 1.0 | 3.0 | 0.0 | 247.0 | 0.0 | 4.0 | 0.0 |
| 19 | 15.0 | 190.0 | 1.0 | 202.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 3.0 | 0.0 | 8.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 20 | 4.0 | 9.0 | 1.0 | 1.0 | 65.0 | 104.0 | 4.0 | 20.0 | 1.0 | 0.0 | 0.0 | 0.0 | 100.0 | 102.0 | 3.0 | 6.0 |
| 21 | 27.0 | 24.0 | 105.0 | 14.0 | 203.0 | 2.0 | 6.0 | 4.0 | 1.0 | 1.0 | 4.0 | 2.0 | 10.0 | 1.0 | 13.0 | 3.0 |
| 22 | 63.0 | 27.0 | 17.0 | 134.0 | 0.0 | 0.0 | 1.0 | 27.0 | 21.0 | 3.0 | 5.0 | 99.0 | 5.0 | 1.0 | 0.0 | 17.0 |
| 23 | 2.0 | 2.0 | 2.0 | 83.0 | 0.0 | 1.0 | 0.0 | 30.0 | 0.0 | 1.0 | 1.0 | 21.0 | 3.0 | 5.0 | 6.0 | 263.0 |
| 24 | 1.0 | 0.0 | 4.0 | 0.0 | 2.0 | 0.0 | 7.0 | 0.0 | 1.0 | 0.0 | 7.0 | 1.0 | 28.0 | 1.0 | 361.0 | 7.0 |
| 25 | 4.0 | 0.0 | 28.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 46.0 | 2.0 | 326.0 | 5.0 | 0.0 | 1.0 | 7.0 | 0.0 |
| 26 | 8.0 | 1.0 | 41.0 | 1.0 | 1.0 | 0.0 | 0.0 | 2.0 | 43.0 | 2.0 | 273.0 | 43.0 | 0.0 | 0.0 | 5.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -4.256 | -2.962 | -2.962 | -0.268 | -4.256 | -4.256 | -4.256 | -2.065 | -2.962 | -1.805 | -1.429 | 2.659 | -4.256 | -4.256 | -2.416 | -2.416 |
| 02 | -4.256 | -4.256 | -4.256 | -2.962 | -4.256 | -2.065 | -4.256 | -2.416 | -4.256 | -1.429 | -4.256 | -2.065 | -1.429 | 2.257 | -2.416 | 1.684 |
| 03 | -4.256 | -4.256 | -4.256 | -1.429 | -4.256 | -2.962 | -4.256 | 2.288 | -4.256 | -4.256 | -4.256 | -2.416 | -4.256 | -2.962 | -4.256 | 1.718 |
| 04 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -2.416 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -2.416 | 2.742 | -2.065 | -4.256 |
| 05 | -4.256 | -4.256 | -4.256 | -2.416 | -1.805 | -0.616 | -4.256 | 2.703 | -4.256 | -4.256 | -4.256 | -2.065 | -4.256 | -4.256 | -4.256 | -4.256 |
| 06 | -4.256 | -4.256 | -2.962 | -2.065 | -4.256 | -4.256 | -4.256 | -0.616 | -4.256 | -4.256 | -4.256 | -4.256 | -2.416 | -2.962 | -1.429 | 2.693 |
| 07 | -4.256 | -4.256 | -2.416 | -4.256 | -4.256 | -4.256 | -2.962 | -4.256 | -4.256 | -4.256 | -1.284 | -4.256 | -2.416 | -2.416 | 2.723 | -2.962 |
| 08 | -2.962 | -4.256 | -4.256 | -2.962 | -4.256 | -4.256 | -4.256 | -2.416 | 2.072 | -1.284 | -1.429 | 1.972 | -4.256 | -4.256 | -4.256 | -2.962 |
| 09 | 2.038 | -2.962 | -1.805 | -2.065 | -1.284 | -4.256 | -4.256 | -4.256 | -1.429 | -4.256 | -4.256 | -4.256 | 1.978 | -2.962 | -4.256 | -2.416 |
| 10 | 0.441 | -1.805 | 2.571 | -0.486 | -4.256 | -4.256 | -2.962 | -2.962 | -4.256 | -4.256 | -2.065 | -2.962 | -2.962 | -4.256 | -1.805 | -4.256 |
| 11 | -0.549 | -4.256 | 0.028 | -4.256 | -1.805 | -4.256 | -4.256 | -4.256 | 1.919 | -4.256 | 1.862 | -1.805 | -0.549 | -4.256 | -2.065 | -4.256 |
| 12 | -4.256 | 2.09 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | 2.023 | -4.256 | -4.256 | -4.256 | -1.805 | -4.256 | -4.256 |
| 13 | -4.256 | -4.256 | -4.256 | -4.256 | 2.757 | -4.256 | -2.962 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 |
| 14 | 1.265 | -4.256 | 2.503 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -2.962 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 |
| 15 | -4.256 | 1.276 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -2.416 | 2.494 | -2.962 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 |
| 16 | -2.416 | -4.256 | -4.256 | -4.256 | 2.752 | -4.256 | -4.256 | -4.256 | -2.962 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 |
| 17 | -0.22 | 0.164 | 1.483 | 2.245 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 | -4.256 |
| 18 | -0.268 | -4.256 | -2.962 | -4.256 | 0.064 | -4.256 | -2.065 | -4.256 | 1.449 | -2.962 | -2.065 | -4.256 | 2.229 | -4.256 | -1.805 | -4.256 |
| 19 | -0.549 | 1.967 | -2.962 | 2.028 | -4.256 | -4.256 | -4.256 | -2.962 | -4.256 | -2.065 | -4.256 | -1.156 | -4.256 | -4.256 | -4.256 | -4.256 |
| 20 | -1.805 | -1.044 | -2.962 | -2.962 | 0.898 | 1.366 | -1.805 | -0.268 | -2.962 | -4.256 | -4.256 | -4.256 | 1.327 | 1.347 | -2.065 | -1.429 |
| 21 | 0.028 | -0.088 | 1.376 | -0.616 | 2.033 | -2.416 | -1.429 | -1.805 | -2.962 | -2.962 | -1.805 | -2.416 | -0.942 | -2.962 | -0.688 | -2.065 |
| 22 | 0.867 | 0.028 | -0.427 | 1.619 | -4.256 | -4.256 | -2.962 | 0.028 | -0.22 | -2.065 | -1.6 | 1.317 | -1.6 | -2.962 | -4.256 | -0.427 |
| 23 | -2.416 | -2.416 | -2.416 | 1.141 | -4.256 | -2.962 | -4.256 | 0.132 | -4.256 | -2.962 | -2.962 | -0.22 | -2.065 | -1.6 | -1.429 | 2.292 |
| 24 | -2.962 | -4.256 | -1.805 | -4.256 | -2.416 | -4.256 | -1.284 | -4.256 | -2.962 | -4.256 | -1.284 | -2.962 | 0.064 | -2.962 | 2.608 | -1.284 |
| 25 | -1.805 | -4.256 | 0.064 | -4.256 | -2.962 | -4.256 | -4.256 | -4.256 | 0.555 | -2.416 | 2.506 | -1.6 | -4.256 | -2.962 | -1.284 | -4.256 |
| 26 | -1.156 | -2.962 | 0.441 | -2.962 | -2.962 | -4.256 | -4.256 | -2.416 | 0.488 | -2.416 | 2.329 | 0.488 | -4.256 | -4.256 | -1.6 | -4.256 |