Model info
| Transcription factor | ZNF582 (GeneCards) | ||||||||
| Model | ZN582_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO |  | ||||||||
| LOGO (reverse complement) | 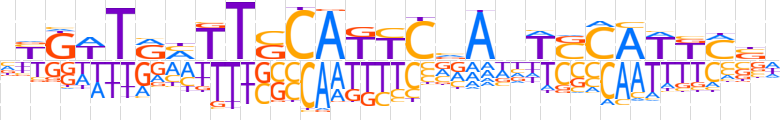 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 27 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | WSKMWTGSWnTbGAATGSAAKYAWCRn | ||||||||
| Best auROC (human) | 0.87 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 3 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 228 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF620-like factors {2.3.3.35} | ||||||||
| HGNC | HGNC:26421 | ||||||||
| EntrezGene | GeneID:147948 (SSTAR profile) | ||||||||
| UniProt ID | ZN582_HUMAN | ||||||||
| UniProt AC | Q96NG8 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF582 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF582 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 3.0 | 35.0 | 35.0 | 1.0 | 7.0 | 2.0 | 0.0 | 0.0 | 5.0 | 2.0 | 2.0 | 1.0 | 15.0 | 77.0 | 34.0 | 7.0 |
| 02 | 3.0 | 2.0 | 24.0 | 1.0 | 13.0 | 0.0 | 65.0 | 38.0 | 2.0 | 1.0 | 68.0 | 0.0 | 0.0 | 0.0 | 9.0 | 0.0 |
| 03 | 13.0 | 1.0 | 3.0 | 1.0 | 2.0 | 0.0 | 1.0 | 0.0 | 142.0 | 3.0 | 20.0 | 1.0 | 1.0 | 36.0 | 1.0 | 1.0 |
| 04 | 145.0 | 0.0 | 7.0 | 6.0 | 3.0 | 0.0 | 1.0 | 36.0 | 20.0 | 2.0 | 3.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 |
| 05 | 0.0 | 0.0 | 2.0 | 169.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 2.0 | 9.0 | 3.0 | 0.0 | 33.0 | 6.0 |
| 06 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 4.0 | 32.0 | 2.0 | 0.0 | 183.0 | 1.0 |
| 07 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 63.0 | 112.0 | 10.0 | 0.0 | 0.0 | 36.0 | 0.0 |
| 08 | 0.0 | 0.0 | 2.0 | 0.0 | 20.0 | 4.0 | 5.0 | 36.0 | 148.0 | 0.0 | 0.0 | 0.0 | 5.0 | 0.0 | 2.0 | 4.0 |
| 09 | 76.0 | 37.0 | 39.0 | 21.0 | 0.0 | 1.0 | 0.0 | 3.0 | 0.0 | 0.0 | 1.0 | 8.0 | 0.0 | 2.0 | 0.0 | 38.0 |
| 10 | 4.0 | 1.0 | 0.0 | 71.0 | 0.0 | 1.0 | 0.0 | 39.0 | 1.0 | 1.0 | 0.0 | 38.0 | 3.0 | 1.0 | 0.0 | 66.0 |
| 11 | 0.0 | 1.0 | 5.0 | 2.0 | 0.0 | 1.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 13.0 | 81.0 | 48.0 | 72.0 |
| 12 | 1.0 | 0.0 | 12.0 | 0.0 | 14.0 | 0.0 | 67.0 | 2.0 | 1.0 | 1.0 | 54.0 | 0.0 | 7.0 | 0.0 | 67.0 | 0.0 |
| 13 | 0.0 | 0.0 | 23.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 150.0 | 0.0 | 50.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 |
| 14 | 152.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 71.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 15 | 0.0 | 7.0 | 0.0 | 146.0 | 3.0 | 0.0 | 1.0 | 68.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 16 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 11.0 | 0.0 | 204.0 | 0.0 |
| 17 | 0.0 | 11.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 102.0 | 112.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 18 | 0.0 | 0.0 | 0.0 | 0.0 | 112.0 | 0.0 | 1.0 | 0.0 | 111.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 19 | 188.0 | 27.0 | 9.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 20 | 12.0 | 0.0 | 66.0 | 111.0 | 26.0 | 0.0 | 0.0 | 2.0 | 4.0 | 0.0 | 4.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 21 | 0.0 | 35.0 | 2.0 | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 9.0 | 57.0 | 4.0 | 106.0 | 4.0 | 0.0 |
| 22 | 2.0 | 0.0 | 2.0 | 0.0 | 143.0 | 0.0 | 1.0 | 1.0 | 12.0 | 2.0 | 1.0 | 0.0 | 61.0 | 0.0 | 0.0 | 1.0 |
| 23 | 119.0 | 4.0 | 3.0 | 92.0 | 2.0 | 0.0 | 0.0 | 0.0 | 2.0 | 1.0 | 1.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 |
| 24 | 12.0 | 92.0 | 11.0 | 9.0 | 4.0 | 2.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 87.0 | 1.0 | 4.0 |
| 25 | 6.0 | 2.0 | 6.0 | 2.0 | 131.0 | 21.0 | 26.0 | 7.0 | 7.0 | 1.0 | 2.0 | 2.0 | 8.0 | 0.0 | 3.0 | 2.0 |
| 26 | 13.0 | 41.0 | 49.0 | 49.0 | 4.0 | 20.0 | 0.0 | 0.0 | 31.0 | 2.0 | 4.0 | 0.0 | 1.0 | 1.0 | 11.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.466 | 0.893 | 0.893 | -2.38 | -0.678 | -1.822 | -3.754 | -3.754 | -0.997 | -1.822 | -1.822 | -2.38 | 0.059 | 1.677 | 0.865 | -0.678 |
| 02 | -1.466 | -1.822 | 0.52 | -2.38 | -0.081 | -3.754 | 1.508 | 0.975 | -1.822 | -2.38 | 1.553 | -3.754 | -3.754 | -3.754 | -0.437 | -3.754 |
| 03 | -0.081 | -2.38 | -1.466 | -2.38 | -1.822 | -3.754 | -2.38 | -3.754 | 2.287 | -1.466 | 0.341 | -2.38 | -2.38 | 0.921 | -2.38 | -2.38 |
| 04 | 2.307 | -3.754 | -0.678 | -0.825 | -1.466 | -3.754 | -2.38 | 0.921 | 0.341 | -1.822 | -1.466 | -3.754 | -1.466 | -3.754 | -3.754 | -3.754 |
| 05 | -3.754 | -3.754 | -1.822 | 2.46 | -3.754 | -3.754 | -3.754 | -1.822 | -3.754 | -3.754 | -1.822 | -0.437 | -1.466 | -3.754 | 0.835 | -0.825 |
| 06 | -3.754 | -3.754 | -3.754 | -1.466 | -3.754 | -3.754 | -3.754 | -3.754 | -2.38 | -3.754 | -1.204 | 0.805 | -1.822 | -3.754 | 2.54 | -2.38 |
| 07 | -3.754 | -1.822 | -3.754 | -2.38 | -3.754 | -3.754 | -3.754 | -3.754 | -1.822 | 1.477 | 2.05 | -0.336 | -3.754 | -3.754 | 0.921 | -3.754 |
| 08 | -3.754 | -3.754 | -1.822 | -3.754 | 0.341 | -1.204 | -0.997 | 0.921 | 2.328 | -3.754 | -3.754 | -3.754 | -0.997 | -3.754 | -1.822 | -1.204 |
| 09 | 1.664 | 0.948 | 1.001 | 0.389 | -3.754 | -2.38 | -3.754 | -1.466 | -3.754 | -3.754 | -2.38 | -0.551 | -3.754 | -1.822 | -3.754 | 0.975 |
| 10 | -1.204 | -2.38 | -3.754 | 1.596 | -3.754 | -2.38 | -3.754 | 1.001 | -2.38 | -2.38 | -3.754 | 0.975 | -1.466 | -2.38 | -3.754 | 1.523 |
| 11 | -3.754 | -2.38 | -0.997 | -1.822 | -3.754 | -2.38 | -1.466 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -0.081 | 1.727 | 1.207 | 1.61 |
| 12 | -2.38 | -3.754 | -0.159 | -3.754 | -0.009 | -3.754 | 1.538 | -1.822 | -2.38 | -2.38 | 1.324 | -3.754 | -0.678 | -3.754 | 1.538 | -3.754 |
| 13 | -3.754 | -3.754 | 0.478 | -3.754 | -3.754 | -3.754 | -3.754 | -2.38 | 2.341 | -3.754 | 1.247 | -3.754 | -1.822 | -3.754 | -3.754 | -3.754 |
| 14 | 2.354 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -2.38 | 1.596 | -3.754 | -2.38 | -3.754 | -2.38 | -3.754 | -3.754 |
| 15 | -3.754 | -0.678 | -3.754 | 2.314 | -1.466 | -3.754 | -2.38 | 1.553 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -2.38 |
| 16 | -3.754 | -3.754 | -1.466 | -3.754 | -3.754 | -3.754 | -0.678 | -3.754 | -3.754 | -3.754 | -2.38 | -3.754 | -0.243 | -3.754 | 2.648 | -3.754 |
| 17 | -3.754 | -0.243 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | 1.957 | 2.05 | -2.38 | -3.754 | -3.754 | -3.754 | -3.754 |
| 18 | -3.754 | -3.754 | -3.754 | -3.754 | 2.05 | -3.754 | -2.38 | -3.754 | 2.041 | -3.754 | -3.754 | -2.38 | -2.38 | -3.754 | -3.754 | -3.754 |
| 19 | 2.567 | 0.637 | -0.437 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -2.38 | -3.754 | -3.754 | -2.38 | -3.754 | -3.754 | -3.754 |
| 20 | -0.159 | -3.754 | 1.523 | 2.041 | 0.599 | -3.754 | -3.754 | -1.822 | -1.204 | -3.754 | -1.204 | -2.38 | -3.754 | -3.754 | -3.754 | -3.754 |
| 21 | -3.754 | 0.893 | -1.822 | -0.997 | -3.754 | -3.754 | -3.754 | -3.754 | -3.754 | -1.204 | -0.437 | 1.377 | -1.204 | 1.995 | -1.204 | -3.754 |
| 22 | -1.822 | -3.754 | -1.822 | -3.754 | 2.294 | -3.754 | -2.38 | -2.38 | -0.159 | -1.822 | -2.38 | -3.754 | 1.445 | -3.754 | -3.754 | -2.38 |
| 23 | 2.11 | -1.204 | -1.466 | 1.854 | -1.822 | -3.754 | -3.754 | -3.754 | -1.822 | -2.38 | -2.38 | -3.754 | -2.38 | -2.38 | -3.754 | -3.754 |
| 24 | -0.159 | 1.854 | -0.243 | -0.437 | -1.204 | -1.822 | -3.754 | -3.754 | -3.754 | -1.204 | -3.754 | -3.754 | -3.754 | 1.798 | -2.38 | -1.204 |
| 25 | -0.825 | -1.822 | -0.825 | -1.822 | 2.206 | 0.389 | 0.599 | -0.678 | -0.678 | -2.38 | -1.822 | -1.822 | -0.551 | -3.754 | -1.466 | -1.822 |
| 26 | -0.081 | 1.05 | 1.227 | 1.227 | -1.204 | 0.341 | -3.754 | -3.754 | 0.773 | -1.822 | -1.204 | -3.754 | -2.38 | -2.38 | -0.243 | -3.754 |