Model info
| Transcription factor | ZNF85 (GeneCards) | ||||||||
| Model | ZNF85_HUMAN.H11DI.0.C | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 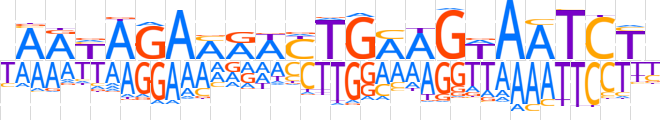 | ||||||||
| LOGO (reverse complement) | 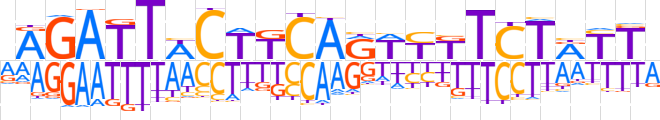 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | C | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | YAAYAGAMRWYTGMAGWAATCTY | ||||||||
| Best auROC (human) | 0.908 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 4 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 199 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | unclassified {2.3.3.0} | ||||||||
| HGNC | HGNC:13160 | ||||||||
| EntrezGene | GeneID:7639 (SSTAR profile) | ||||||||
| UniProt ID | ZNF85_HUMAN | ||||||||
| UniProt AC | Q03923 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZNF85 expression | ||||||||
| ReMap ChIP-seq dataset list | ZNF85 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 6.0 | 4.0 | 3.0 | 3.0 | 27.0 | 3.0 | 0.0 | 1.0 | 4.0 | 2.0 | 1.0 | 1.0 | 133.0 | 3.0 | 5.0 | 3.0 |
| 02 | 146.0 | 2.0 | 17.0 | 5.0 | 6.0 | 2.0 | 1.0 | 3.0 | 5.0 | 0.0 | 4.0 | 0.0 | 3.0 | 1.0 | 1.0 | 3.0 |
| 03 | 15.0 | 15.0 | 6.0 | 124.0 | 2.0 | 1.0 | 0.0 | 2.0 | 3.0 | 4.0 | 5.0 | 11.0 | 0.0 | 2.0 | 3.0 | 6.0 |
| 04 | 17.0 | 0.0 | 1.0 | 2.0 | 19.0 | 0.0 | 0.0 | 3.0 | 10.0 | 0.0 | 1.0 | 3.0 | 132.0 | 1.0 | 7.0 | 3.0 |
| 05 | 17.0 | 2.0 | 156.0 | 3.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 7.0 | 1.0 | 3.0 | 0.0 | 8.0 | 0.0 |
| 06 | 19.0 | 1.0 | 2.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 167.0 | 1.0 | 1.0 | 2.0 | 4.0 | 0.0 | 0.0 | 0.0 |
| 07 | 135.0 | 40.0 | 10.0 | 6.0 | 3.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 |
| 08 | 71.0 | 0.0 | 71.0 | 1.0 | 38.0 | 0.0 | 1.0 | 1.0 | 6.0 | 1.0 | 3.0 | 0.0 | 2.0 | 0.0 | 4.0 | 0.0 |
| 09 | 77.0 | 1.0 | 6.0 | 33.0 | 0.0 | 1.0 | 0.0 | 0.0 | 68.0 | 3.0 | 1.0 | 7.0 | 0.0 | 0.0 | 1.0 | 1.0 |
| 10 | 11.0 | 105.0 | 11.0 | 18.0 | 1.0 | 4.0 | 0.0 | 0.0 | 1.0 | 6.0 | 0.0 | 1.0 | 2.0 | 37.0 | 1.0 | 1.0 |
| 11 | 3.0 | 0.0 | 1.0 | 11.0 | 2.0 | 2.0 | 1.0 | 147.0 | 3.0 | 1.0 | 0.0 | 8.0 | 2.0 | 0.0 | 4.0 | 14.0 |
| 12 | 1.0 | 0.0 | 9.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 4.0 | 0.0 | 14.0 | 0.0 | 166.0 | 0.0 |
| 13 | 19.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 116.0 | 60.0 | 1.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 14 | 121.0 | 5.0 | 4.0 | 5.0 | 39.0 | 0.0 | 2.0 | 19.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 3.0 |
| 15 | 6.0 | 0.0 | 152.0 | 2.0 | 1.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 | 0.0 | 0.0 | 28.0 | 0.0 |
| 16 | 0.0 | 0.0 | 0.0 | 7.0 | 0.0 | 0.0 | 0.0 | 0.0 | 34.0 | 6.0 | 20.0 | 130.0 | 0.0 | 0.0 | 0.0 | 2.0 |
| 17 | 34.0 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 | 0.0 | 0.0 | 20.0 | 0.0 | 0.0 | 0.0 | 139.0 | 0.0 | 0.0 | 0.0 |
| 18 | 168.0 | 29.0 | 1.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 19 | 2.0 | 4.0 | 2.0 | 160.0 | 0.0 | 0.0 | 0.0 | 29.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 20 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 4.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 0.0 | 3.0 | 177.0 | 3.0 | 8.0 |
| 21 | 1.0 | 1.0 | 0.0 | 2.0 | 12.0 | 19.0 | 1.0 | 151.0 | 0.0 | 0.0 | 0.0 | 3.0 | 1.0 | 1.0 | 0.0 | 7.0 |
| 22 | 4.0 | 3.0 | 4.0 | 3.0 | 4.0 | 9.0 | 0.0 | 8.0 | 0.0 | 0.0 | 0.0 | 1.0 | 4.0 | 48.0 | 10.0 | 101.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.702 | -1.081 | -1.344 | -1.344 | 0.761 | -1.344 | -3.653 | -2.261 | -1.081 | -1.701 | -2.261 | -2.261 | 2.346 | -1.344 | -0.873 | -1.344 |
| 02 | 2.439 | -1.701 | 0.306 | -0.873 | -0.702 | -1.701 | -2.261 | -1.344 | -0.873 | -3.653 | -1.081 | -3.653 | -1.344 | -2.261 | -2.261 | -1.344 |
| 03 | 0.183 | 0.183 | -0.702 | 2.276 | -1.701 | -2.261 | -3.653 | -1.701 | -1.344 | -1.081 | -0.873 | -0.119 | -3.653 | -1.701 | -1.344 | -0.702 |
| 04 | 0.306 | -3.653 | -2.261 | -1.701 | 0.415 | -3.653 | -3.653 | -1.344 | -0.212 | -3.653 | -2.261 | -1.344 | 2.338 | -2.261 | -0.555 | -1.344 |
| 05 | 0.306 | -1.701 | 2.505 | -1.344 | -2.261 | -3.653 | -3.653 | -3.653 | -2.261 | -3.653 | -0.555 | -2.261 | -1.344 | -3.653 | -0.427 | -3.653 |
| 06 | 0.415 | -2.261 | -1.701 | -3.653 | -2.261 | -2.261 | -3.653 | -3.653 | 2.573 | -2.261 | -2.261 | -1.701 | -1.081 | -3.653 | -3.653 | -3.653 |
| 07 | 2.361 | 1.15 | -0.212 | -0.702 | -1.344 | -3.653 | -3.653 | -3.653 | -1.344 | -3.653 | -3.653 | -3.653 | -1.701 | -3.653 | -3.653 | -3.653 |
| 08 | 1.72 | -3.653 | 1.72 | -2.261 | 1.099 | -3.653 | -2.261 | -2.261 | -0.702 | -2.261 | -1.344 | -3.653 | -1.701 | -3.653 | -1.081 | -3.653 |
| 09 | 1.801 | -2.261 | -0.702 | 0.96 | -3.653 | -2.261 | -3.653 | -3.653 | 1.677 | -1.344 | -2.261 | -0.555 | -3.653 | -3.653 | -2.261 | -2.261 |
| 10 | -0.119 | 2.11 | -0.119 | 0.362 | -2.261 | -1.081 | -3.653 | -3.653 | -2.261 | -0.702 | -3.653 | -2.261 | -1.701 | 1.073 | -2.261 | -2.261 |
| 11 | -1.344 | -3.653 | -2.261 | -0.119 | -1.701 | -1.701 | -2.261 | 2.446 | -1.344 | -2.261 | -3.653 | -0.427 | -1.701 | -3.653 | -1.081 | 0.115 |
| 12 | -2.261 | -3.653 | -0.314 | -3.653 | -1.701 | -3.653 | -2.261 | -3.653 | -1.701 | -3.653 | -1.081 | -3.653 | 0.115 | -3.653 | 2.567 | -3.653 |
| 13 | 0.415 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | 2.209 | 1.553 | -2.261 | -1.344 | -3.653 | -3.653 | -3.653 | -3.653 |
| 14 | 2.252 | -0.873 | -1.081 | -0.873 | 1.125 | -3.653 | -1.701 | 0.415 | -3.653 | -3.653 | -3.653 | -2.261 | -3.653 | -3.653 | -3.653 | -1.344 |
| 15 | -0.702 | -3.653 | 2.479 | -1.701 | -2.261 | -3.653 | -1.081 | -3.653 | -3.653 | -3.653 | -0.702 | -3.653 | -3.653 | -3.653 | 0.797 | -3.653 |
| 16 | -3.653 | -3.653 | -3.653 | -0.555 | -3.653 | -3.653 | -3.653 | -3.653 | 0.989 | -0.702 | 0.465 | 2.323 | -3.653 | -3.653 | -3.653 | -1.701 |
| 17 | 0.989 | -3.653 | -3.653 | -3.653 | -0.702 | -3.653 | -3.653 | -3.653 | 0.465 | -3.653 | -3.653 | -3.653 | 2.39 | -3.653 | -3.653 | -3.653 |
| 18 | 2.579 | 0.832 | -2.261 | -2.261 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 | -3.653 |
| 19 | -1.701 | -1.081 | -1.701 | 2.53 | -3.653 | -3.653 | -3.653 | 0.832 | -3.653 | -3.653 | -3.653 | -2.261 | -3.653 | -3.653 | -3.653 | -2.261 |
| 20 | -3.653 | -2.261 | -3.653 | -2.261 | -3.653 | -1.081 | -3.653 | -3.653 | -2.261 | -2.261 | -3.653 | -3.653 | -1.344 | 2.631 | -1.344 | -0.427 |
| 21 | -2.261 | -2.261 | -3.653 | -1.701 | -0.035 | 0.415 | -2.261 | 2.473 | -3.653 | -3.653 | -3.653 | -1.344 | -2.261 | -2.261 | -3.653 | -0.555 |
| 22 | -1.081 | -1.344 | -1.081 | -1.344 | -1.081 | -0.314 | -3.653 | -0.427 | -3.653 | -3.653 | -3.653 | -2.261 | -1.081 | 1.331 | -0.212 | 2.071 |