Model info
| Transcription factor | Ascl1 | ||||||||
| Model | ASCL1_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO |  | ||||||||
| LOGO (reverse complement) | 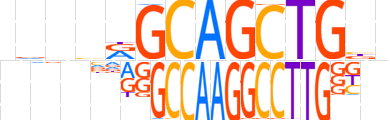 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 14 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nvCAGCTGCYbnbn | ||||||||
| Best auROC (human) | 0.976 | ||||||||
| Best auROC (mouse) | 0.993 | ||||||||
| Peak sets in benchmark (human) | 30 | ||||||||
| Peak sets in benchmark (mouse) | 25 | ||||||||
| Aligned words | 342 | ||||||||
| TF family | MyoD / ASC-related factors {1.2.2} | ||||||||
| TF subfamily | Achaete-Scute-like factors {1.2.2.2} | ||||||||
| MGI | MGI:96919 | ||||||||
| EntrezGene | GeneID:17172 (SSTAR profile) | ||||||||
| UniProt ID | ASCL1_MOUSE | ||||||||
| UniProt AC | Q02067 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Ascl1 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 23.0 | 16.0 | 16.0 | 1.0 | 42.0 | 39.0 | 17.0 | 7.0 | 32.0 | 51.0 | 17.0 | 1.0 | 9.0 | 22.0 | 45.0 | 3.0 |
| 02 | 0.0 | 106.0 | 0.0 | 0.0 | 0.0 | 128.0 | 0.0 | 0.0 | 0.0 | 95.0 | 0.0 | 0.0 | 0.0 | 12.0 | 0.0 | 0.0 |
| 03 | 0.0 | 0.0 | 0.0 | 0.0 | 341.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 04 | 0.0 | 8.0 | 333.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 05 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 8.0 | 0.0 | 0.0 | 0.0 | 333.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 06 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 341.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 07 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 341.0 | 0.0 |
| 08 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 333.0 | 0.0 | 8.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 09 | 0.0 | 0.0 | 0.0 | 0.0 | 45.0 | 131.0 | 1.0 | 156.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 6.0 | 0.0 | 2.0 |
| 10 | 1.0 | 2.0 | 33.0 | 9.0 | 28.0 | 43.0 | 9.0 | 57.0 | 0.0 | 1.0 | 0.0 | 0.0 | 10.0 | 54.0 | 77.0 | 17.0 |
| 11 | 8.0 | 9.0 | 17.0 | 5.0 | 26.0 | 37.0 | 10.0 | 27.0 | 12.0 | 46.0 | 43.0 | 18.0 | 5.0 | 26.0 | 35.0 | 17.0 |
| 12 | 4.0 | 10.0 | 23.0 | 14.0 | 10.0 | 35.0 | 3.0 | 70.0 | 16.0 | 30.0 | 40.0 | 19.0 | 5.0 | 19.0 | 19.0 | 24.0 |
| 13 | 8.0 | 7.0 | 9.0 | 11.0 | 16.0 | 32.0 | 16.0 | 30.0 | 21.0 | 26.0 | 19.0 | 19.0 | 15.0 | 22.0 | 58.0 | 32.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.075 | -0.281 | -0.281 | -2.765 | 0.67 | 0.597 | -0.222 | -1.08 | 0.401 | 0.863 | -0.222 | -2.765 | -0.839 | 0.031 | 0.738 | -1.863 |
| 02 | -4.086 | 1.591 | -4.086 | -4.086 | -4.086 | 1.779 | -4.086 | -4.086 | -4.086 | 1.481 | -4.086 | -4.086 | -4.086 | -0.561 | -4.086 | -4.086 |
| 03 | -4.086 | -4.086 | -4.086 | -4.086 | 2.757 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 |
| 04 | -4.086 | -0.952 | 2.733 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 |
| 05 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -0.952 | -4.086 | -4.086 | -4.086 | 2.733 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 |
| 06 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | 2.757 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 |
| 07 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | 2.757 | -4.086 |
| 08 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | 2.733 | -4.086 | -0.952 | -4.086 | -4.086 | -4.086 | -4.086 |
| 09 | -4.086 | -4.086 | -4.086 | -4.086 | 0.738 | 1.802 | -2.765 | 1.976 | -4.086 | -4.086 | -4.086 | -4.086 | -4.086 | -1.226 | -4.086 | -2.216 |
| 10 | -2.765 | -2.216 | 0.431 | -0.839 | 0.269 | 0.693 | -0.839 | 0.973 | -4.086 | -2.765 | -4.086 | -4.086 | -0.738 | 0.919 | 1.272 | -0.222 |
| 11 | -0.952 | -0.839 | -0.222 | -1.396 | 0.196 | 0.544 | -0.738 | 0.233 | -0.561 | 0.76 | 0.693 | -0.166 | -1.396 | 0.196 | 0.489 | -0.222 |
| 12 | -1.603 | -0.738 | 0.075 | -0.411 | -0.738 | 0.489 | -1.863 | 1.177 | -0.281 | 0.337 | 0.622 | -0.113 | -1.396 | -0.113 | -0.113 | 0.117 |
| 13 | -0.952 | -1.08 | -0.839 | -0.646 | -0.281 | 0.401 | -0.281 | 0.337 | -0.015 | 0.196 | -0.113 | -0.113 | -0.344 | 0.031 | 0.99 | 0.401 |