Model info
| Transcription factor | Tp53 | ||||||||
| Model | P53_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 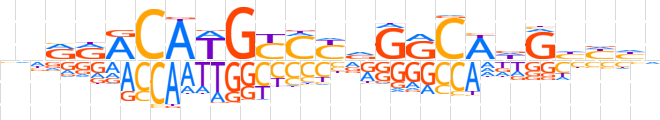 | ||||||||
| LOGO (reverse complement) | 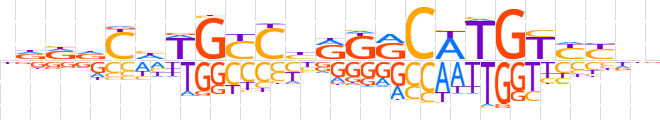 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 23 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | vnRRACAWGYMYdGRCWdGhYhn | ||||||||
| Best auROC (human) | 0.999 | ||||||||
| Best auROC (mouse) | 0.994 | ||||||||
| Peak sets in benchmark (human) | 141 | ||||||||
| Peak sets in benchmark (mouse) | 39 | ||||||||
| Aligned words | 514 | ||||||||
| TF family | p53-related factors {6.3.1} | ||||||||
| TF subfamily | p53 {6.3.1.0.1} | ||||||||
| MGI | MGI:98834 | ||||||||
| EntrezGene | GeneID:22059 (SSTAR profile) | ||||||||
| UniProt ID | P53_MOUSE | ||||||||
| UniProt AC | P02340 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Tp53 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 32.0 | 40.0 | 33.0 | 12.0 | 125.0 | 30.0 | 11.0 | 47.0 | 19.0 | 18.0 | 22.0 | 32.0 | 20.0 | 18.0 | 22.0 | 12.0 |
| 02 | 34.0 | 2.0 | 156.0 | 4.0 | 64.0 | 18.0 | 14.0 | 10.0 | 31.0 | 3.0 | 51.0 | 3.0 | 24.0 | 9.0 | 67.0 | 3.0 |
| 03 | 34.0 | 5.0 | 98.0 | 16.0 | 12.0 | 1.0 | 6.0 | 13.0 | 64.0 | 10.0 | 201.0 | 13.0 | 4.0 | 6.0 | 7.0 | 3.0 |
| 04 | 98.0 | 3.0 | 11.0 | 2.0 | 18.0 | 1.0 | 2.0 | 1.0 | 215.0 | 1.0 | 96.0 | 0.0 | 29.0 | 1.0 | 11.0 | 4.0 |
| 05 | 5.0 | 346.0 | 2.0 | 7.0 | 1.0 | 4.0 | 1.0 | 0.0 | 0.0 | 118.0 | 1.0 | 1.0 | 0.0 | 7.0 | 0.0 | 0.0 |
| 06 | 4.0 | 1.0 | 1.0 | 0.0 | 432.0 | 0.0 | 18.0 | 25.0 | 2.0 | 1.0 | 1.0 | 0.0 | 5.0 | 1.0 | 2.0 | 0.0 |
| 07 | 96.0 | 2.0 | 13.0 | 332.0 | 0.0 | 0.0 | 0.0 | 3.0 | 7.0 | 0.0 | 5.0 | 10.0 | 10.0 | 0.0 | 0.0 | 15.0 |
| 08 | 1.0 | 0.0 | 112.0 | 0.0 | 1.0 | 0.0 | 1.0 | 0.0 | 2.0 | 2.0 | 13.0 | 1.0 | 1.0 | 4.0 | 354.0 | 1.0 |
| 09 | 1.0 | 3.0 | 0.0 | 1.0 | 0.0 | 2.0 | 0.0 | 4.0 | 15.0 | 298.0 | 2.0 | 165.0 | 0.0 | 0.0 | 0.0 | 2.0 |
| 10 | 4.0 | 8.0 | 1.0 | 3.0 | 16.0 | 273.0 | 2.0 | 12.0 | 0.0 | 0.0 | 1.0 | 1.0 | 25.0 | 112.0 | 14.0 | 21.0 |
| 11 | 10.0 | 22.0 | 9.0 | 4.0 | 19.0 | 275.0 | 3.0 | 96.0 | 4.0 | 11.0 | 1.0 | 2.0 | 4.0 | 20.0 | 5.0 | 8.0 |
| 12 | 12.0 | 1.0 | 21.0 | 3.0 | 148.0 | 30.0 | 121.0 | 29.0 | 10.0 | 0.0 | 7.0 | 1.0 | 17.0 | 16.0 | 62.0 | 15.0 |
| 13 | 14.0 | 1.0 | 166.0 | 6.0 | 28.0 | 0.0 | 9.0 | 10.0 | 12.0 | 1.0 | 197.0 | 1.0 | 6.0 | 0.0 | 42.0 | 0.0 |
| 14 | 32.0 | 1.0 | 25.0 | 2.0 | 1.0 | 0.0 | 1.0 | 0.0 | 93.0 | 6.0 | 306.0 | 9.0 | 9.0 | 1.0 | 5.0 | 2.0 |
| 15 | 0.0 | 127.0 | 7.0 | 1.0 | 1.0 | 7.0 | 0.0 | 0.0 | 12.0 | 313.0 | 7.0 | 5.0 | 0.0 | 5.0 | 6.0 | 2.0 |
| 16 | 2.0 | 8.0 | 1.0 | 2.0 | 375.0 | 19.0 | 9.0 | 49.0 | 4.0 | 12.0 | 1.0 | 3.0 | 0.0 | 2.0 | 3.0 | 3.0 |
| 17 | 104.0 | 11.0 | 105.0 | 161.0 | 10.0 | 5.0 | 4.0 | 22.0 | 6.0 | 2.0 | 3.0 | 3.0 | 18.0 | 0.0 | 12.0 | 27.0 |
| 18 | 12.0 | 7.0 | 115.0 | 4.0 | 10.0 | 5.0 | 2.0 | 1.0 | 13.0 | 6.0 | 99.0 | 6.0 | 5.0 | 7.0 | 191.0 | 10.0 |
| 19 | 8.0 | 11.0 | 14.0 | 7.0 | 9.0 | 4.0 | 0.0 | 12.0 | 56.0 | 243.0 | 19.0 | 89.0 | 3.0 | 1.0 | 15.0 | 2.0 |
| 20 | 13.0 | 31.0 | 19.0 | 13.0 | 37.0 | 194.0 | 5.0 | 23.0 | 7.0 | 21.0 | 6.0 | 14.0 | 11.0 | 57.0 | 22.0 | 20.0 |
| 21 | 14.0 | 26.0 | 17.0 | 11.0 | 47.0 | 193.0 | 6.0 | 57.0 | 9.0 | 18.0 | 7.0 | 18.0 | 9.0 | 29.0 | 13.0 | 19.0 |
| 22 | 15.0 | 19.0 | 36.0 | 9.0 | 137.0 | 42.0 | 49.0 | 38.0 | 7.0 | 13.0 | 9.0 | 14.0 | 21.0 | 38.0 | 35.0 | 11.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.037 | 0.258 | 0.068 | -0.924 | 1.391 | -0.026 | -1.008 | 0.418 | -0.476 | -0.529 | -0.332 | 0.037 | -0.425 | -0.529 | -0.332 | -0.924 |
| 02 | 0.097 | -2.57 | 1.612 | -1.962 | 0.725 | -0.529 | -0.774 | -1.1 | 0.006 | -2.22 | 0.499 | -2.22 | -0.246 | -1.201 | 0.77 | -2.22 |
| 03 | 0.097 | -1.756 | 1.148 | -0.644 | -0.924 | -3.113 | -1.586 | -0.846 | 0.725 | -1.1 | 1.865 | -0.846 | -1.962 | -1.586 | -1.441 | -2.22 |
| 04 | 1.148 | -2.22 | -1.008 | -2.57 | -0.529 | -3.113 | -2.57 | -3.113 | 1.932 | -3.113 | 1.128 | -4.388 | -0.06 | -3.113 | -1.008 | -1.962 |
| 05 | -1.756 | 2.407 | -2.57 | -1.441 | -3.113 | -1.962 | -3.113 | -4.388 | -4.388 | 1.334 | -3.113 | -3.113 | -4.388 | -1.441 | -4.388 | -4.388 |
| 06 | -1.962 | -3.113 | -3.113 | -4.388 | 2.629 | -4.388 | -0.529 | -0.206 | -2.57 | -3.113 | -3.113 | -4.388 | -1.756 | -3.113 | -2.57 | -4.388 |
| 07 | 1.128 | -2.57 | -0.846 | 2.366 | -4.388 | -4.388 | -4.388 | -2.22 | -1.441 | -4.388 | -1.756 | -1.1 | -1.1 | -4.388 | -4.388 | -0.707 |
| 08 | -3.113 | -4.388 | 1.282 | -4.388 | -3.113 | -4.388 | -3.113 | -4.388 | -2.57 | -2.57 | -0.846 | -3.113 | -3.113 | -1.962 | 2.43 | -3.113 |
| 09 | -3.113 | -2.22 | -4.388 | -3.113 | -4.388 | -2.57 | -4.388 | -1.962 | -0.707 | 2.258 | -2.57 | 1.668 | -4.388 | -4.388 | -4.388 | -2.57 |
| 10 | -1.962 | -1.314 | -3.113 | -2.22 | -0.644 | 2.17 | -2.57 | -0.924 | -4.388 | -4.388 | -3.113 | -3.113 | -0.206 | 1.282 | -0.774 | -0.378 |
| 11 | -1.1 | -0.332 | -1.201 | -1.962 | -0.476 | 2.178 | -2.22 | 1.128 | -1.962 | -1.008 | -3.113 | -2.57 | -1.962 | -0.425 | -1.756 | -1.314 |
| 12 | -0.924 | -3.113 | -0.378 | -2.22 | 1.559 | -0.026 | 1.359 | -0.06 | -1.1 | -4.388 | -1.441 | -3.113 | -0.585 | -0.644 | 0.693 | -0.707 |
| 13 | -0.774 | -3.113 | 1.674 | -1.586 | -0.094 | -4.388 | -1.201 | -1.1 | -0.924 | -3.113 | 1.845 | -3.113 | -1.586 | -4.388 | 0.306 | -4.388 |
| 14 | 0.037 | -3.113 | -0.206 | -2.57 | -3.113 | -4.388 | -3.113 | -4.388 | 1.096 | -1.586 | 2.284 | -1.201 | -1.201 | -3.113 | -1.756 | -2.57 |
| 15 | -4.388 | 1.407 | -1.441 | -3.113 | -3.113 | -1.441 | -4.388 | -4.388 | -0.924 | 2.307 | -1.441 | -1.756 | -4.388 | -1.756 | -1.586 | -2.57 |
| 16 | -2.57 | -1.314 | -3.113 | -2.57 | 2.488 | -0.476 | -1.201 | 0.459 | -1.962 | -0.924 | -3.113 | -2.22 | -4.388 | -2.57 | -2.22 | -2.22 |
| 17 | 1.208 | -1.008 | 1.217 | 1.643 | -1.1 | -1.756 | -1.962 | -0.332 | -1.586 | -2.57 | -2.22 | -2.22 | -0.529 | -4.388 | -0.924 | -0.13 |
| 18 | -0.924 | -1.441 | 1.308 | -1.962 | -1.1 | -1.756 | -2.57 | -3.113 | -0.846 | -1.586 | 1.159 | -1.586 | -1.756 | -1.441 | 1.814 | -1.1 |
| 19 | -1.314 | -1.008 | -0.774 | -1.441 | -1.201 | -1.962 | -4.388 | -0.924 | 0.592 | 2.054 | -0.476 | 1.053 | -2.22 | -3.113 | -0.707 | -2.57 |
| 20 | -0.846 | 0.006 | -0.476 | -0.846 | 0.181 | 1.829 | -1.756 | -0.288 | -1.441 | -0.378 | -1.586 | -0.774 | -1.008 | 0.609 | -0.332 | -0.425 |
| 21 | -0.774 | -0.168 | -0.585 | -1.008 | 0.418 | 1.824 | -1.586 | 0.609 | -1.201 | -0.529 | -1.441 | -0.529 | -1.201 | -0.06 | -0.846 | -0.476 |
| 22 | -0.707 | -0.476 | 0.154 | -1.201 | 1.482 | 0.306 | 0.459 | 0.207 | -1.441 | -0.846 | -1.201 | -0.774 | -0.378 | 0.207 | 0.126 | -1.008 |