Model info
| Transcription factor | Prdm16 | ||||||||
| Model | PRD16_MOUSE.H11DI.0.B | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 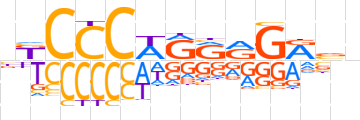 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 13 | ||||||||
| Quality | B | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nKCCCWRRRGRvn | ||||||||
| Best auROC (human) | |||||||||
| Best auROC (mouse) | 0.876 | ||||||||
| Peak sets in benchmark (human) | |||||||||
| Peak sets in benchmark (mouse) | 3 | ||||||||
| Aligned words | 501 | ||||||||
| TF family | Factors with multiple dispersed zinc fingers {2.3.4} | ||||||||
| TF subfamily | Evi-1-like factors {2.3.4.14} | ||||||||
| MGI | MGI:1917923 | ||||||||
| EntrezGene | GeneID:70673 (SSTAR profile) | ||||||||
| UniProt ID | PRD16_MOUSE | ||||||||
| UniProt AC | A2A935 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Prdm16 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 11.0 | 12.0 | 20.0 | 50.0 | 10.0 | 7.0 | 10.0 | 98.0 | 10.0 | 21.0 | 31.0 | 86.0 | 5.0 | 12.0 | 25.0 | 92.0 |
| 02 | 0.0 | 36.0 | 0.0 | 0.0 | 2.0 | 48.0 | 0.0 | 2.0 | 1.0 | 83.0 | 0.0 | 2.0 | 5.0 | 317.0 | 4.0 | 0.0 |
| 03 | 1.0 | 6.0 | 0.0 | 1.0 | 4.0 | 413.0 | 1.0 | 66.0 | 0.0 | 2.0 | 1.0 | 1.0 | 0.0 | 3.0 | 0.0 | 1.0 |
| 04 | 0.0 | 5.0 | 0.0 | 0.0 | 7.0 | 411.0 | 3.0 | 3.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 68.0 | 0.0 | 1.0 |
| 05 | 4.0 | 0.0 | 0.0 | 3.0 | 269.0 | 4.0 | 13.0 | 200.0 | 2.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 4.0 |
| 06 | 76.0 | 0.0 | 176.0 | 23.0 | 0.0 | 0.0 | 4.0 | 0.0 | 1.0 | 0.0 | 12.0 | 0.0 | 22.0 | 6.0 | 169.0 | 11.0 |
| 07 | 14.0 | 7.0 | 77.0 | 1.0 | 2.0 | 0.0 | 1.0 | 3.0 | 45.0 | 33.0 | 250.0 | 33.0 | 5.0 | 4.0 | 23.0 | 2.0 |
| 08 | 9.0 | 2.0 | 52.0 | 3.0 | 25.0 | 1.0 | 13.0 | 5.0 | 164.0 | 10.0 | 171.0 | 6.0 | 5.0 | 1.0 | 25.0 | 8.0 |
| 09 | 3.0 | 28.0 | 170.0 | 2.0 | 2.0 | 3.0 | 9.0 | 0.0 | 4.0 | 22.0 | 234.0 | 1.0 | 0.0 | 4.0 | 16.0 | 2.0 |
| 10 | 1.0 | 1.0 | 5.0 | 2.0 | 41.0 | 3.0 | 4.0 | 9.0 | 289.0 | 16.0 | 104.0 | 20.0 | 3.0 | 1.0 | 1.0 | 0.0 |
| 11 | 52.0 | 150.0 | 96.0 | 36.0 | 5.0 | 9.0 | 3.0 | 4.0 | 38.0 | 50.0 | 22.0 | 4.0 | 3.0 | 17.0 | 9.0 | 2.0 |
| 12 | 27.0 | 17.0 | 28.0 | 26.0 | 67.0 | 81.0 | 13.0 | 65.0 | 40.0 | 28.0 | 28.0 | 34.0 | 7.0 | 10.0 | 19.0 | 10.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -1.022 | -0.938 | -0.439 | 0.465 | -1.114 | -1.454 | -1.114 | 1.135 | -1.114 | -0.392 | -0.008 | 1.004 | -1.77 | -0.938 | -0.22 | 1.072 |
| 02 | -4.4 | 0.14 | -4.4 | -4.4 | -2.584 | 0.425 | -4.4 | -2.584 | -3.126 | 0.969 | -4.4 | -2.584 | -1.77 | 2.306 | -1.975 | -4.4 |
| 03 | -3.126 | -1.6 | -4.4 | -3.126 | -1.975 | 2.57 | -3.126 | 0.741 | -4.4 | -2.584 | -3.126 | -3.126 | -4.4 | -2.234 | -4.4 | -3.126 |
| 04 | -4.4 | -1.77 | -4.4 | -4.4 | -1.454 | 2.565 | -2.234 | -2.234 | -4.4 | -2.584 | -4.4 | -4.4 | -4.4 | 0.771 | -4.4 | -3.126 |
| 05 | -1.975 | -4.4 | -4.4 | -2.234 | 2.142 | -1.975 | -0.86 | 1.846 | -2.584 | -4.4 | -4.4 | -3.126 | -4.4 | -4.4 | -4.4 | -1.975 |
| 06 | 0.881 | -4.4 | 1.718 | -0.302 | -4.4 | -4.4 | -1.975 | -4.4 | -3.126 | -4.4 | -0.938 | -4.4 | -0.346 | -1.6 | 1.678 | -1.022 |
| 07 | -0.788 | -1.454 | 0.894 | -3.126 | -2.584 | -4.4 | -3.126 | -2.234 | 0.361 | 0.054 | 2.069 | 0.054 | -1.77 | -1.975 | -0.302 | -2.584 |
| 08 | -1.215 | -2.584 | 0.504 | -2.234 | -0.22 | -3.126 | -0.86 | -1.77 | 1.648 | -1.114 | 1.69 | -1.6 | -1.77 | -3.126 | -0.22 | -1.328 |
| 09 | -2.234 | -0.108 | 1.684 | -2.584 | -2.584 | -2.234 | -1.215 | -4.4 | -1.975 | -0.346 | 2.003 | -3.126 | -4.4 | -1.975 | -0.658 | -2.584 |
| 10 | -3.126 | -3.126 | -1.77 | -2.584 | 0.269 | -2.234 | -1.975 | -1.215 | 2.213 | -0.658 | 1.194 | -0.439 | -2.234 | -3.126 | -3.126 | -4.4 |
| 11 | 0.504 | 1.559 | 1.114 | 0.14 | -1.77 | -1.215 | -2.234 | -1.975 | 0.193 | 0.465 | -0.346 | -1.975 | -2.234 | -0.599 | -1.215 | -2.584 |
| 12 | -0.144 | -0.599 | -0.108 | -0.181 | 0.756 | 0.945 | -0.86 | 0.726 | 0.244 | -0.108 | -0.108 | 0.083 | -1.454 | -1.114 | -0.49 | -1.114 |