Model info
| Transcription factor | STAT5B (GeneCards) | ||||||||
| Model | STA5B_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 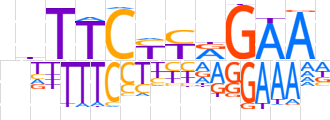 | ||||||||
| LOGO (reverse complement) | 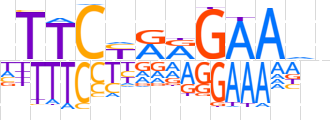 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 12 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | dhTTCYTRGAAn | ||||||||
| Best auROC (human) | 0.963 | ||||||||
| Best auROC (mouse) | 0.963 | ||||||||
| Peak sets in benchmark (human) | 16 | ||||||||
| Peak sets in benchmark (mouse) | 31 | ||||||||
| Aligned words | 508 | ||||||||
| TF family | STAT factors {6.2.1} | ||||||||
| TF subfamily | STAT5B {6.2.1.0.6} | ||||||||
| HGNC | HGNC:11367 | ||||||||
| EntrezGene | GeneID:6777 (SSTAR profile) | ||||||||
| UniProt ID | STA5B_HUMAN | ||||||||
| UniProt AC | P51692 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | STAT5B expression | ||||||||
| ReMap ChIP-seq dataset list | STAT5B datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 32.0 | 38.0 | 39.0 | 76.0 | 9.0 | 13.0 | 4.0 | 40.0 | 40.0 | 28.0 | 19.0 | 36.0 | 14.0 | 19.0 | 27.0 | 66.0 |
| 02 | 1.0 | 1.0 | 1.0 | 92.0 | 1.0 | 0.0 | 1.0 | 96.0 | 0.0 | 4.0 | 1.0 | 84.0 | 3.0 | 1.0 | 3.0 | 211.0 |
| 03 | 1.0 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 6.0 | 2.0 | 0.0 | 0.0 | 4.0 | 42.0 | 9.0 | 2.0 | 430.0 |
| 04 | 1.0 | 43.0 | 1.0 | 0.0 | 0.0 | 8.0 | 0.0 | 1.0 | 1.0 | 1.0 | 0.0 | 0.0 | 13.0 | 428.0 | 3.0 | 0.0 |
| 05 | 1.0 | 8.0 | 1.0 | 5.0 | 39.0 | 127.0 | 7.0 | 307.0 | 0.0 | 2.0 | 0.0 | 2.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 06 | 0.0 | 24.0 | 0.0 | 16.0 | 0.0 | 40.0 | 0.0 | 98.0 | 0.0 | 6.0 | 0.0 | 2.0 | 0.0 | 172.0 | 0.0 | 142.0 |
| 07 | 0.0 | 0.0 | 0.0 | 0.0 | 174.0 | 6.0 | 25.0 | 37.0 | 0.0 | 0.0 | 0.0 | 0.0 | 113.0 | 0.0 | 123.0 | 22.0 |
| 08 | 0.0 | 0.0 | 284.0 | 3.0 | 0.0 | 0.0 | 6.0 | 0.0 | 0.0 | 0.0 | 140.0 | 8.0 | 0.0 | 0.0 | 59.0 | 0.0 |
| 09 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 426.0 | 1.0 | 5.0 | 57.0 | 8.0 | 0.0 | 0.0 | 3.0 |
| 10 | 412.0 | 5.0 | 14.0 | 3.0 | 1.0 | 0.0 | 0.0 | 0.0 | 4.0 | 1.0 | 0.0 | 0.0 | 57.0 | 0.0 | 3.0 | 0.0 |
| 11 | 179.0 | 76.0 | 96.0 | 123.0 | 1.0 | 0.0 | 3.0 | 2.0 | 3.0 | 5.0 | 3.0 | 6.0 | 0.0 | 1.0 | 2.0 | 0.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.023 | 0.193 | 0.219 | 0.881 | -1.215 | -0.86 | -1.975 | 0.244 | 0.244 | -0.108 | -0.49 | 0.14 | -0.788 | -0.49 | -0.144 | 0.741 |
| 02 | -3.126 | -3.126 | -3.126 | 1.072 | -3.126 | -4.4 | -3.126 | 1.114 | -4.4 | -1.975 | -3.126 | 0.981 | -2.234 | -3.126 | -2.234 | 1.899 |
| 03 | -3.126 | -4.4 | -4.4 | -1.975 | -4.4 | -4.4 | -4.4 | -1.6 | -2.584 | -4.4 | -4.4 | -1.975 | 0.293 | -1.215 | -2.584 | 2.61 |
| 04 | -3.126 | 0.316 | -3.126 | -4.4 | -4.4 | -1.328 | -4.4 | -3.126 | -3.126 | -3.126 | -4.4 | -4.4 | -0.86 | 2.606 | -2.234 | -4.4 |
| 05 | -3.126 | -1.328 | -3.126 | -1.77 | 0.219 | 1.393 | -1.454 | 2.274 | -4.4 | -2.584 | -4.4 | -2.584 | -4.4 | -3.126 | -4.4 | -4.4 |
| 06 | -4.4 | -0.26 | -4.4 | -0.658 | -4.4 | 0.244 | -4.4 | 1.135 | -4.4 | -1.6 | -4.4 | -2.584 | -4.4 | 1.695 | -4.4 | 1.504 |
| 07 | -4.4 | -4.4 | -4.4 | -4.4 | 1.707 | -1.6 | -0.22 | 0.167 | -4.4 | -4.4 | -4.4 | -4.4 | 1.276 | -4.4 | 1.361 | -0.346 |
| 08 | -4.4 | -4.4 | 2.196 | -2.234 | -4.4 | -4.4 | -1.6 | -4.4 | -4.4 | -4.4 | 1.49 | -1.328 | -4.4 | -4.4 | 0.63 | -4.4 |
| 09 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | 2.601 | -3.126 | -1.77 | 0.595 | -1.328 | -4.4 | -4.4 | -2.234 |
| 10 | 2.568 | -1.77 | -0.788 | -2.234 | -3.126 | -4.4 | -4.4 | -4.4 | -1.975 | -3.126 | -4.4 | -4.4 | 0.595 | -4.4 | -2.234 | -4.4 |
| 11 | 1.735 | 0.881 | 1.114 | 1.361 | -3.126 | -4.4 | -2.234 | -2.584 | -2.234 | -1.77 | -2.234 | -1.6 | -4.4 | -3.126 | -2.584 | -4.4 |