Model info
| Transcription factor | STAT2 (GeneCards) | ||||||||
| Model | STAT2_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO |  | ||||||||
| LOGO (reverse complement) | 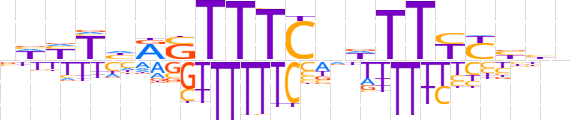 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 20 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | ndvRRAAhnGAAASYdRRWn | ||||||||
| Best auROC (human) | 0.982 | ||||||||
| Best auROC (mouse) | 0.995 | ||||||||
| Peak sets in benchmark (human) | 11 | ||||||||
| Peak sets in benchmark (mouse) | 8 | ||||||||
| Aligned words | 500 | ||||||||
| TF family | STAT factors {6.2.1} | ||||||||
| TF subfamily | STAT2 {6.2.1.0.2} | ||||||||
| HGNC | HGNC:11363 | ||||||||
| EntrezGene | GeneID:6773 (SSTAR profile) | ||||||||
| UniProt ID | STAT2_HUMAN | ||||||||
| UniProt AC | P52630 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | STAT2 expression | ||||||||
| ReMap ChIP-seq dataset list | STAT2 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 66.0 | 23.0 | 50.0 | 13.0 | 81.0 | 16.0 | 10.0 | 26.0 | 31.0 | 10.0 | 44.0 | 11.0 | 24.0 | 14.0 | 57.0 | 24.0 |
| 02 | 69.0 | 23.0 | 99.0 | 11.0 | 36.0 | 11.0 | 6.0 | 10.0 | 76.0 | 11.0 | 69.0 | 5.0 | 25.0 | 13.0 | 29.0 | 7.0 |
| 03 | 48.0 | 1.0 | 151.0 | 6.0 | 41.0 | 2.0 | 7.0 | 8.0 | 50.0 | 3.0 | 144.0 | 6.0 | 6.0 | 1.0 | 24.0 | 2.0 |
| 04 | 32.0 | 0.0 | 108.0 | 5.0 | 4.0 | 0.0 | 3.0 | 0.0 | 239.0 | 0.0 | 83.0 | 4.0 | 8.0 | 0.0 | 14.0 | 0.0 |
| 05 | 281.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 207.0 | 0.0 | 0.0 | 1.0 | 9.0 | 0.0 | 0.0 | 0.0 |
| 06 | 478.0 | 6.0 | 6.0 | 7.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 07 | 261.0 | 82.0 | 29.0 | 108.0 | 5.0 | 2.0 | 0.0 | 0.0 | 1.0 | 3.0 | 0.0 | 2.0 | 2.0 | 1.0 | 2.0 | 2.0 |
| 08 | 40.0 | 72.0 | 36.0 | 121.0 | 39.0 | 8.0 | 3.0 | 38.0 | 11.0 | 2.0 | 3.0 | 15.0 | 30.0 | 29.0 | 34.0 | 19.0 |
| 09 | 5.0 | 1.0 | 114.0 | 0.0 | 31.0 | 0.0 | 79.0 | 1.0 | 12.0 | 0.0 | 63.0 | 1.0 | 5.0 | 2.0 | 186.0 | 0.0 |
| 10 | 45.0 | 1.0 | 6.0 | 1.0 | 3.0 | 0.0 | 0.0 | 0.0 | 432.0 | 4.0 | 4.0 | 2.0 | 1.0 | 1.0 | 0.0 | 0.0 |
| 11 | 477.0 | 0.0 | 4.0 | 0.0 | 6.0 | 0.0 | 0.0 | 0.0 | 10.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 |
| 12 | 495.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 13 | 23.0 | 321.0 | 149.0 | 6.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 |
| 14 | 7.0 | 3.0 | 0.0 | 14.0 | 31.0 | 59.0 | 2.0 | 229.0 | 16.0 | 31.0 | 5.0 | 97.0 | 0.0 | 3.0 | 0.0 | 3.0 |
| 15 | 7.0 | 1.0 | 42.0 | 4.0 | 40.0 | 8.0 | 24.0 | 24.0 | 2.0 | 0.0 | 5.0 | 0.0 | 64.0 | 29.0 | 185.0 | 65.0 |
| 16 | 83.0 | 9.0 | 15.0 | 6.0 | 26.0 | 3.0 | 2.0 | 7.0 | 222.0 | 10.0 | 14.0 | 10.0 | 68.0 | 6.0 | 10.0 | 9.0 |
| 17 | 280.0 | 24.0 | 62.0 | 33.0 | 14.0 | 5.0 | 0.0 | 9.0 | 19.0 | 6.0 | 13.0 | 3.0 | 7.0 | 8.0 | 11.0 | 6.0 |
| 18 | 226.0 | 26.0 | 41.0 | 27.0 | 16.0 | 12.0 | 4.0 | 11.0 | 45.0 | 10.0 | 11.0 | 20.0 | 16.0 | 10.0 | 11.0 | 14.0 |
| 19 | 52.0 | 115.0 | 112.0 | 24.0 | 23.0 | 8.0 | 4.0 | 23.0 | 20.0 | 20.0 | 16.0 | 11.0 | 6.0 | 17.0 | 30.0 | 19.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.741 | -0.302 | 0.465 | -0.86 | 0.945 | -0.658 | -1.114 | -0.181 | -0.008 | -1.114 | 0.339 | -1.022 | -0.26 | -0.788 | 0.595 | -0.26 |
| 02 | 0.785 | -0.302 | 1.145 | -1.022 | 0.14 | -1.022 | -1.6 | -1.114 | 0.881 | -1.022 | 0.785 | -1.77 | -0.22 | -0.86 | -0.074 | -1.454 |
| 03 | 0.425 | -3.126 | 1.565 | -1.6 | 0.269 | -2.584 | -1.454 | -1.328 | 0.465 | -2.234 | 1.518 | -1.6 | -1.6 | -3.126 | -0.26 | -2.584 |
| 04 | 0.023 | -4.4 | 1.231 | -1.77 | -1.975 | -4.4 | -2.234 | -4.4 | 2.024 | -4.4 | 0.969 | -1.975 | -1.328 | -4.4 | -0.788 | -4.4 |
| 05 | 2.185 | -2.584 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | 1.88 | -4.4 | -4.4 | -3.126 | -1.215 | -4.4 | -4.4 | -4.4 |
| 06 | 2.716 | -1.6 | -1.6 | -1.454 | -2.584 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -3.126 | -4.4 | -4.4 |
| 07 | 2.112 | 0.957 | -0.074 | 1.231 | -1.77 | -2.584 | -4.4 | -4.4 | -3.126 | -2.234 | -4.4 | -2.584 | -2.584 | -3.126 | -2.584 | -2.584 |
| 08 | 0.244 | 0.828 | 0.14 | 1.345 | 0.219 | -1.328 | -2.234 | 0.193 | -1.022 | -2.584 | -2.234 | -0.721 | -0.04 | -0.074 | 0.083 | -0.49 |
| 09 | -1.77 | -3.126 | 1.285 | -4.4 | -0.008 | -4.4 | 0.92 | -3.126 | -0.938 | -4.4 | 0.695 | -3.126 | -1.77 | -2.584 | 1.773 | -4.4 |
| 10 | 0.361 | -3.126 | -1.6 | -3.126 | -2.234 | -4.4 | -4.4 | -4.4 | 2.615 | -1.975 | -1.975 | -2.584 | -3.126 | -3.126 | -4.4 | -4.4 |
| 11 | 2.714 | -4.4 | -1.975 | -4.4 | -1.6 | -4.4 | -4.4 | -4.4 | -1.114 | -4.4 | -4.4 | -4.4 | -2.234 | -4.4 | -4.4 | -4.4 |
| 12 | 2.751 | -4.4 | -4.4 | -3.126 | -4.4 | -4.4 | -4.4 | -4.4 | -1.975 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 |
| 13 | -0.302 | 2.318 | 1.552 | -1.6 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -4.4 | -3.126 | -4.4 | -4.4 | -4.4 |
| 14 | -1.454 | -2.234 | -4.4 | -0.788 | -0.008 | 0.63 | -2.584 | 1.981 | -0.658 | -0.008 | -1.77 | 1.124 | -4.4 | -2.234 | -4.4 | -2.234 |
| 15 | -1.454 | -3.126 | 0.293 | -1.975 | 0.244 | -1.328 | -0.26 | -0.26 | -2.584 | -4.4 | -1.77 | -4.4 | 0.711 | -0.074 | 1.768 | 0.726 |
| 16 | 0.969 | -1.215 | -0.721 | -1.6 | -0.181 | -2.234 | -2.584 | -1.454 | 1.95 | -1.114 | -0.788 | -1.114 | 0.771 | -1.6 | -1.114 | -1.215 |
| 17 | 2.182 | -0.26 | 0.679 | 0.054 | -0.788 | -1.77 | -4.4 | -1.215 | -0.49 | -1.6 | -0.86 | -2.234 | -1.454 | -1.328 | -1.022 | -1.6 |
| 18 | 1.968 | -0.181 | 0.269 | -0.144 | -0.658 | -0.938 | -1.975 | -1.022 | 0.361 | -1.114 | -1.022 | -0.439 | -0.658 | -1.114 | -1.022 | -0.788 |
| 19 | 0.504 | 1.294 | 1.268 | -0.26 | -0.302 | -1.328 | -1.975 | -0.302 | -0.439 | -0.439 | -0.658 | -1.022 | -1.6 | -0.599 | -0.04 | -0.49 |