Model info
| Transcription factor | Nr5a1 | ||||||||
| Model | STF1_MOUSE.H11DI.0.B | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 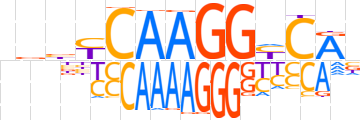 | ||||||||
| LOGO (reverse complement) |  | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 13 | ||||||||
| Quality | B | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nbbYCAAGGYCRn | ||||||||
| Best auROC (human) | 0.775 | ||||||||
| Best auROC (mouse) | 0.908 | ||||||||
| Peak sets in benchmark (human) | 1 | ||||||||
| Peak sets in benchmark (mouse) | 2 | ||||||||
| Aligned words | 500 | ||||||||
| TF family | FTZ-F1-related receptors (NR5) {2.1.5} | ||||||||
| TF subfamily | FTZ-F1 (SF-1) (NR5A1) {2.1.5.0.1} | ||||||||
| MGI | MGI:1346833 | ||||||||
| EntrezGene | GeneID:26423 (SSTAR profile) | ||||||||
| UniProt ID | STF1_MOUSE | ||||||||
| UniProt AC | P33242 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Nr5a1 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 30.0 | 21.0 | 101.0 | 18.0 | 31.0 | 24.0 | 26.0 | 23.0 | 13.0 | 17.0 | 56.0 | 18.0 | 9.0 | 33.0 | 50.0 | 30.0 |
| 02 | 4.0 | 26.0 | 31.0 | 22.0 | 16.0 | 34.0 | 8.0 | 37.0 | 9.0 | 64.0 | 79.0 | 81.0 | 4.0 | 33.0 | 19.0 | 33.0 |
| 03 | 2.0 | 19.0 | 1.0 | 11.0 | 3.0 | 78.0 | 1.0 | 75.0 | 11.0 | 71.0 | 1.0 | 54.0 | 7.0 | 56.0 | 1.0 | 109.0 |
| 04 | 0.0 | 23.0 | 0.0 | 0.0 | 0.0 | 218.0 | 3.0 | 3.0 | 0.0 | 4.0 | 0.0 | 0.0 | 1.0 | 239.0 | 8.0 | 1.0 |
| 05 | 1.0 | 0.0 | 0.0 | 0.0 | 465.0 | 4.0 | 9.0 | 6.0 | 9.0 | 0.0 | 1.0 | 1.0 | 3.0 | 1.0 | 0.0 | 0.0 |
| 06 | 457.0 | 0.0 | 19.0 | 2.0 | 4.0 | 0.0 | 1.0 | 0.0 | 8.0 | 2.0 | 0.0 | 0.0 | 4.0 | 0.0 | 2.0 | 1.0 |
| 07 | 3.0 | 1.0 | 469.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 1.0 | 0.0 | 21.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 |
| 08 | 0.0 | 1.0 | 3.0 | 0.0 | 0.0 | 1.0 | 1.0 | 0.0 | 3.0 | 2.0 | 488.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 09 | 2.0 | 0.0 | 0.0 | 1.0 | 2.0 | 2.0 | 0.0 | 0.0 | 61.0 | 197.0 | 7.0 | 227.0 | 0.0 | 1.0 | 0.0 | 0.0 |
| 10 | 4.0 | 56.0 | 1.0 | 4.0 | 1.0 | 184.0 | 2.0 | 13.0 | 0.0 | 7.0 | 0.0 | 0.0 | 2.0 | 211.0 | 4.0 | 11.0 |
| 11 | 4.0 | 2.0 | 1.0 | 0.0 | 375.0 | 9.0 | 59.0 | 15.0 | 5.0 | 0.0 | 1.0 | 1.0 | 9.0 | 5.0 | 11.0 | 3.0 |
| 12 | 59.0 | 129.0 | 125.0 | 80.0 | 5.0 | 5.0 | 1.0 | 5.0 | 14.0 | 25.0 | 24.0 | 9.0 | 1.0 | 4.0 | 9.0 | 5.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.04 | -0.392 | 1.165 | -0.543 | -0.008 | -0.26 | -0.181 | -0.302 | -0.86 | -0.599 | 0.578 | -0.543 | -1.215 | 0.054 | 0.465 | -0.04 |
| 02 | -1.975 | -0.181 | -0.008 | -0.346 | -0.658 | 0.083 | -1.328 | 0.167 | -1.215 | 0.711 | 0.92 | 0.945 | -1.975 | 0.054 | -0.49 | 0.054 |
| 03 | -2.584 | -0.49 | -3.126 | -1.022 | -2.234 | 0.907 | -3.126 | 0.868 | -1.022 | 0.814 | -3.126 | 0.542 | -1.454 | 0.578 | -3.126 | 1.241 |
| 04 | -4.4 | -0.302 | -4.4 | -4.4 | -4.4 | 1.932 | -2.234 | -2.234 | -4.4 | -1.975 | -4.4 | -4.4 | -3.126 | 2.024 | -1.328 | -3.126 |
| 05 | -3.126 | -4.4 | -4.4 | -4.4 | 2.689 | -1.975 | -1.215 | -1.6 | -1.215 | -4.4 | -3.126 | -3.126 | -2.234 | -3.126 | -4.4 | -4.4 |
| 06 | 2.671 | -4.4 | -0.49 | -2.584 | -1.975 | -4.4 | -3.126 | -4.4 | -1.328 | -2.584 | -4.4 | -4.4 | -1.975 | -4.4 | -2.584 | -3.126 |
| 07 | -2.234 | -3.126 | 2.697 | -4.4 | -4.4 | -3.126 | -3.126 | -4.4 | -3.126 | -4.4 | -0.392 | -4.4 | -4.4 | -4.4 | -2.234 | -4.4 |
| 08 | -4.4 | -3.126 | -2.234 | -4.4 | -4.4 | -3.126 | -3.126 | -4.4 | -2.234 | -2.584 | 2.737 | -3.126 | -4.4 | -4.4 | -4.4 | -4.4 |
| 09 | -2.584 | -4.4 | -4.4 | -3.126 | -2.584 | -2.584 | -4.4 | -4.4 | 0.663 | 1.831 | -1.454 | 1.972 | -4.4 | -3.126 | -4.4 | -4.4 |
| 10 | -1.975 | 0.578 | -3.126 | -1.975 | -3.126 | 1.763 | -2.584 | -0.86 | -4.4 | -1.454 | -4.4 | -4.4 | -2.584 | 1.899 | -1.975 | -1.022 |
| 11 | -1.975 | -2.584 | -3.126 | -4.4 | 2.474 | -1.215 | 0.63 | -0.721 | -1.77 | -4.4 | -3.126 | -3.126 | -1.215 | -1.77 | -1.022 | -2.234 |
| 12 | 0.63 | 1.408 | 1.377 | 0.932 | -1.77 | -1.77 | -3.126 | -1.77 | -0.788 | -0.22 | -0.26 | -1.215 | -3.126 | -1.975 | -1.215 | -1.77 |