Model info
| Transcription factor | TWIST1 (GeneCards) | ||||||||
| Model | TWST1_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 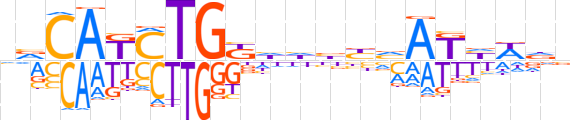 | ||||||||
| LOGO (reverse complement) | 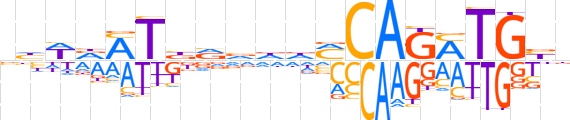 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 20 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | nvCAKCTGKhhbbvAKhWdn | ||||||||
| Best auROC (human) | 0.997 | ||||||||
| Best auROC (mouse) | 0.568 | ||||||||
| Peak sets in benchmark (human) | 9 | ||||||||
| Peak sets in benchmark (mouse) | 8 | ||||||||
| Aligned words | 225 | ||||||||
| TF family | Tal-related factors {1.2.3} | ||||||||
| TF subfamily | Twist-like factors {1.2.3.2} | ||||||||
| HGNC | HGNC:12428 | ||||||||
| EntrezGene | GeneID:7291 (SSTAR profile) | ||||||||
| UniProt ID | TWST1_HUMAN | ||||||||
| UniProt AC | Q15672 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | TWIST1 expression | ||||||||
| ReMap ChIP-seq dataset list | TWIST1 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 37.0 | 14.0 | 17.0 | 1.0 | 36.0 | 12.0 | 9.0 | 3.0 | 28.0 | 14.0 | 14.0 | 2.0 | 8.0 | 11.0 | 17.0 | 2.0 |
| 02 | 2.0 | 105.0 | 2.0 | 0.0 | 5.0 | 46.0 | 0.0 | 0.0 | 6.0 | 50.0 | 1.0 | 0.0 | 0.0 | 8.0 | 0.0 | 0.0 |
| 03 | 11.0 | 0.0 | 2.0 | 0.0 | 204.0 | 0.0 | 4.0 | 1.0 | 0.0 | 0.0 | 3.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 04 | 1.0 | 10.0 | 60.0 | 144.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 7.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 05 | 0.0 | 1.0 | 0.0 | 0.0 | 3.0 | 7.0 | 0.0 | 0.0 | 12.0 | 49.0 | 2.0 | 0.0 | 12.0 | 126.0 | 11.0 | 2.0 |
| 06 | 1.0 | 0.0 | 0.0 | 26.0 | 0.0 | 0.0 | 0.0 | 183.0 | 0.0 | 0.0 | 0.0 | 13.0 | 0.0 | 0.0 | 0.0 | 2.0 |
| 07 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 222.0 | 1.0 |
| 08 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 31.0 | 129.0 | 62.0 | 0.0 | 0.0 | 0.0 | 1.0 |
| 09 | 1.0 | 0.0 | 0.0 | 0.0 | 7.0 | 1.0 | 3.0 | 20.0 | 49.0 | 24.0 | 13.0 | 43.0 | 3.0 | 8.0 | 3.0 | 50.0 |
| 10 | 27.0 | 6.0 | 12.0 | 15.0 | 7.0 | 10.0 | 1.0 | 15.0 | 9.0 | 4.0 | 2.0 | 4.0 | 9.0 | 18.0 | 6.0 | 80.0 |
| 11 | 11.0 | 10.0 | 16.0 | 15.0 | 4.0 | 11.0 | 3.0 | 20.0 | 7.0 | 6.0 | 4.0 | 4.0 | 2.0 | 34.0 | 10.0 | 68.0 |
| 12 | 3.0 | 13.0 | 1.0 | 7.0 | 6.0 | 34.0 | 0.0 | 21.0 | 0.0 | 22.0 | 4.0 | 7.0 | 5.0 | 43.0 | 18.0 | 41.0 |
| 13 | 6.0 | 7.0 | 1.0 | 0.0 | 47.0 | 57.0 | 5.0 | 3.0 | 1.0 | 15.0 | 2.0 | 5.0 | 25.0 | 21.0 | 24.0 | 6.0 |
| 14 | 73.0 | 1.0 | 5.0 | 0.0 | 98.0 | 0.0 | 0.0 | 2.0 | 27.0 | 1.0 | 3.0 | 1.0 | 6.0 | 0.0 | 8.0 | 0.0 |
| 15 | 1.0 | 11.0 | 39.0 | 153.0 | 0.0 | 0.0 | 0.0 | 2.0 | 2.0 | 2.0 | 2.0 | 10.0 | 0.0 | 0.0 | 1.0 | 2.0 |
| 16 | 2.0 | 1.0 | 0.0 | 0.0 | 6.0 | 1.0 | 6.0 | 0.0 | 17.0 | 11.0 | 10.0 | 4.0 | 12.0 | 26.0 | 6.0 | 123.0 |
| 17 | 10.0 | 2.0 | 5.0 | 20.0 | 21.0 | 2.0 | 0.0 | 16.0 | 7.0 | 5.0 | 3.0 | 7.0 | 94.0 | 1.0 | 5.0 | 27.0 |
| 18 | 43.0 | 13.0 | 53.0 | 23.0 | 2.0 | 2.0 | 5.0 | 1.0 | 6.0 | 3.0 | 3.0 | 1.0 | 13.0 | 2.0 | 45.0 | 10.0 |
| 19 | 15.0 | 4.0 | 34.0 | 11.0 | 4.0 | 8.0 | 1.0 | 7.0 | 9.0 | 41.0 | 33.0 | 23.0 | 8.0 | 13.0 | 8.0 | 6.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.953 | -0.004 | 0.186 | -2.376 | 0.926 | -0.155 | -0.433 | -1.462 | 0.677 | -0.004 | -0.004 | -1.818 | -0.546 | -0.239 | 0.186 | -1.818 |
| 02 | -1.818 | 1.99 | -1.818 | -3.751 | -0.992 | 1.169 | -3.751 | -3.751 | -0.821 | 1.251 | -2.376 | -3.751 | -3.751 | -0.546 | -3.751 | -3.751 |
| 03 | -0.239 | -3.751 | -1.818 | -3.751 | 2.652 | -3.751 | -1.2 | -2.376 | -3.751 | -3.751 | -1.462 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 |
| 04 | -2.376 | -0.331 | 1.433 | 2.305 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 | -1.818 | -0.674 | -3.751 | -3.751 | -2.376 | -3.751 |
| 05 | -3.751 | -2.376 | -3.751 | -3.751 | -1.462 | -0.674 | -3.751 | -3.751 | -0.155 | 1.231 | -1.818 | -3.751 | -0.155 | 2.172 | -0.239 | -1.818 |
| 06 | -2.376 | -3.751 | -3.751 | 0.604 | -3.751 | -3.751 | -3.751 | 2.544 | -3.751 | -3.751 | -3.751 | -0.077 | -3.751 | -3.751 | -3.751 | -1.818 |
| 07 | -3.751 | -3.751 | -2.376 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 | -3.751 | -2.376 | -3.751 | 2.737 | -2.376 |
| 08 | -3.751 | -3.751 | -3.751 | -2.376 | -3.751 | -3.751 | -3.751 | -3.751 | -2.376 | 0.778 | 2.195 | 1.465 | -3.751 | -3.751 | -3.751 | -2.376 |
| 09 | -2.376 | -3.751 | -3.751 | -3.751 | -0.674 | -2.376 | -1.462 | 0.345 | 1.231 | 0.525 | -0.077 | 1.102 | -1.462 | -0.546 | -1.462 | 1.251 |
| 10 | 0.641 | -0.821 | -0.155 | 0.063 | -0.674 | -0.331 | -2.376 | 0.063 | -0.433 | -1.2 | -1.818 | -1.2 | -0.433 | 0.242 | -0.821 | 1.719 |
| 11 | -0.239 | -0.331 | 0.126 | 0.063 | -1.2 | -0.239 | -1.462 | 0.345 | -0.674 | -0.821 | -1.2 | -1.2 | -1.818 | 0.869 | -0.331 | 1.557 |
| 12 | -1.462 | -0.077 | -2.376 | -0.674 | -0.821 | 0.869 | -3.751 | 0.393 | -3.751 | 0.439 | -1.2 | -0.674 | -0.992 | 1.102 | 0.242 | 1.054 |
| 13 | -0.821 | -0.674 | -2.376 | -3.751 | 1.19 | 1.382 | -0.992 | -1.462 | -2.376 | 0.063 | -1.818 | -0.992 | 0.565 | 0.393 | 0.525 | -0.821 |
| 14 | 1.628 | -2.376 | -0.992 | -3.751 | 1.921 | -3.751 | -3.751 | -1.818 | 0.641 | -2.376 | -1.462 | -2.376 | -0.821 | -3.751 | -0.546 | -3.751 |
| 15 | -2.376 | -0.239 | 1.005 | 2.365 | -3.751 | -3.751 | -3.751 | -1.818 | -1.818 | -1.818 | -1.818 | -0.331 | -3.751 | -3.751 | -2.376 | -1.818 |
| 16 | -1.818 | -2.376 | -3.751 | -3.751 | -0.821 | -2.376 | -0.821 | -3.751 | 0.186 | -0.239 | -0.331 | -1.2 | -0.155 | 0.604 | -0.821 | 2.148 |
| 17 | -0.331 | -1.818 | -0.992 | 0.345 | 0.393 | -1.818 | -3.751 | 0.126 | -0.674 | -0.992 | -1.462 | -0.674 | 1.88 | -2.376 | -0.992 | 0.641 |
| 18 | 1.102 | -0.077 | 1.309 | 0.483 | -1.818 | -1.818 | -0.992 | -2.376 | -0.821 | -1.462 | -1.462 | -2.376 | -0.077 | -1.818 | 1.147 | -0.331 |
| 19 | 0.063 | -1.2 | 0.869 | -0.239 | -1.2 | -0.546 | -2.376 | -0.674 | -0.433 | 1.054 | 0.839 | 0.483 | -0.546 | -0.077 | -0.546 | -0.821 |