Model info
| Transcription factor | ZEB1 (GeneCards) | ||||||||
| Model | ZEB1_HUMAN.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 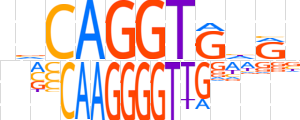 | ||||||||
| LOGO (reverse complement) | 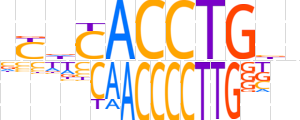 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 11 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | bvCAGGTGhRb | ||||||||
| Best auROC (human) | 0.951 | ||||||||
| Best auROC (mouse) | |||||||||
| Peak sets in benchmark (human) | 12 | ||||||||
| Peak sets in benchmark (mouse) | |||||||||
| Aligned words | 644 | ||||||||
| TF family | HD-ZF factors {3.1.8} | ||||||||
| TF subfamily | ZEB {3.1.8.3} | ||||||||
| HGNC | HGNC:11642 | ||||||||
| EntrezGene | GeneID:6935 (SSTAR profile) | ||||||||
| UniProt ID | ZEB1_HUMAN | ||||||||
| UniProt AC | P37275 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | ZEB1 expression | ||||||||
| ReMap ChIP-seq dataset list | ZEB1 datasets | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 17.0 | 13.0 | 25.0 | 1.0 | 75.0 | 48.0 | 37.0 | 27.0 | 68.0 | 65.0 | 28.0 | 7.0 | 17.0 | 18.0 | 39.0 | 7.0 |
| 02 | 3.0 | 172.0 | 1.0 | 1.0 | 1.0 | 143.0 | 0.0 | 0.0 | 0.0 | 128.0 | 0.0 | 1.0 | 0.0 | 41.0 | 1.0 | 0.0 |
| 03 | 4.0 | 0.0 | 0.0 | 0.0 | 484.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 |
| 04 | 1.0 | 0.0 | 491.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 05 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 491.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 06 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 492.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 07 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 105.0 | 0.0 | 383.0 | 4.0 |
| 08 | 53.0 | 13.0 | 30.0 | 9.0 | 0.0 | 0.0 | 0.0 | 0.0 | 190.0 | 66.0 | 30.0 | 97.0 | 3.0 | 0.0 | 0.0 | 1.0 |
| 09 | 38.0 | 19.0 | 184.0 | 5.0 | 19.0 | 8.0 | 46.0 | 6.0 | 16.0 | 7.0 | 37.0 | 0.0 | 11.0 | 6.0 | 87.0 | 3.0 |
| 10 | 14.0 | 24.0 | 34.0 | 12.0 | 7.0 | 7.0 | 5.0 | 21.0 | 40.0 | 106.0 | 142.0 | 66.0 | 4.0 | 2.0 | 5.0 | 3.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.583 | -0.844 | -0.204 | -3.111 | 0.884 | 0.441 | 0.183 | -0.128 | 0.787 | 0.742 | -0.092 | -1.439 | -0.583 | -0.527 | 0.235 | -1.439 |
| 02 | -2.218 | 1.711 | -3.111 | -3.111 | -3.111 | 1.527 | -4.387 | -4.387 | -4.387 | 1.417 | -4.387 | -3.111 | -4.387 | 0.285 | -3.111 | -4.387 |
| 03 | -1.96 | -4.387 | -4.387 | -4.387 | 2.744 | -4.387 | -4.387 | -4.387 | -2.568 | -4.387 | -4.387 | -4.387 | -2.568 | -4.387 | -4.387 | -4.387 |
| 04 | -3.111 | -4.387 | 2.759 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 |
| 05 | -4.387 | -4.387 | -3.111 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | 2.759 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 |
| 06 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | 2.761 | -4.387 | -4.387 | -4.387 | -4.387 |
| 07 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | -4.387 | 1.219 | -4.387 | 2.511 | -1.96 |
| 08 | 0.539 | -0.844 | -0.024 | -1.199 | -4.387 | -4.387 | -4.387 | -4.387 | 1.811 | 0.757 | -0.024 | 1.14 | -2.218 | -4.387 | -4.387 | -3.111 |
| 09 | 0.209 | -0.474 | 1.779 | -1.754 | -0.474 | -1.312 | 0.399 | -1.584 | -0.642 | -1.439 | 0.183 | -4.387 | -1.006 | -1.584 | 1.032 | -2.218 |
| 10 | -0.772 | -0.244 | 0.099 | -0.922 | -1.439 | -1.439 | -1.754 | -0.376 | 0.26 | 1.229 | 1.52 | 0.757 | -1.96 | -2.568 | -1.754 | -2.218 |