Model info
| Transcription factor | Klf5 | ||||||||
| Model | KLF5_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 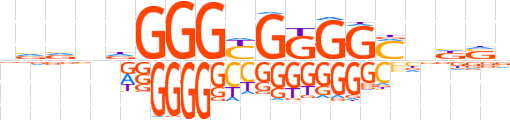 | ||||||||
| LOGO (reverse complement) | 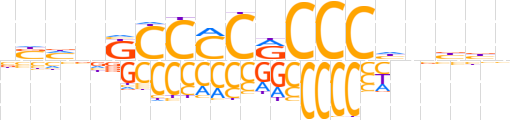 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 18 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | vvvvdGGGYGGGGMndSv | ||||||||
| Best auROC (human) | 0.96 | ||||||||
| Best auROC (mouse) | 0.988 | ||||||||
| Peak sets in benchmark (human) | 32 | ||||||||
| Peak sets in benchmark (mouse) | 23 | ||||||||
| Aligned words | 504 | ||||||||
| TF family | Three-zinc finger Krüppel-related factors {2.3.1} | ||||||||
| TF subfamily | Krüppel-like factors {2.3.1.2} | ||||||||
| MGI | MGI:1338056 | ||||||||
| EntrezGene | GeneID:12224 (SSTAR profile) | ||||||||
| UniProt ID | KLF5_MOUSE | ||||||||
| UniProt AC | Q9Z0Z7 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Klf5 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 19.0 | 14.0 | 51.0 | 7.0 | 36.0 | 29.0 | 40.0 | 12.0 | 64.0 | 50.0 | 99.0 | 18.0 | 6.0 | 9.0 | 37.0 | 7.0 |
| 02 | 30.0 | 10.0 | 70.0 | 15.0 | 25.0 | 22.0 | 43.0 | 12.0 | 37.0 | 44.0 | 127.0 | 19.0 | 5.0 | 10.0 | 22.0 | 7.0 |
| 03 | 20.0 | 21.0 | 42.0 | 14.0 | 24.0 | 23.0 | 9.0 | 30.0 | 63.0 | 90.0 | 95.0 | 14.0 | 6.0 | 16.0 | 27.0 | 4.0 |
| 04 | 39.0 | 1.0 | 64.0 | 9.0 | 49.0 | 8.0 | 54.0 | 39.0 | 60.0 | 11.0 | 34.0 | 68.0 | 14.0 | 1.0 | 40.0 | 7.0 |
| 05 | 4.0 | 0.0 | 158.0 | 0.0 | 1.0 | 0.0 | 20.0 | 0.0 | 2.0 | 2.0 | 187.0 | 1.0 | 2.0 | 1.0 | 120.0 | 0.0 |
| 06 | 0.0 | 0.0 | 9.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 1.0 | 0.0 | 481.0 | 3.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 07 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 2.0 | 2.0 | 490.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 |
| 08 | 0.0 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 2.0 | 56.0 | 311.0 | 0.0 | 127.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 09 | 8.0 | 0.0 | 48.0 | 0.0 | 13.0 | 1.0 | 292.0 | 5.0 | 0.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 126.0 | 2.0 |
| 10 | 0.0 | 0.0 | 22.0 | 2.0 | 0.0 | 0.0 | 0.0 | 1.0 | 12.0 | 0.0 | 332.0 | 122.0 | 0.0 | 0.0 | 7.0 | 0.0 |
| 11 | 0.0 | 0.0 | 12.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 36.0 | 0.0 | 320.0 | 5.0 | 2.0 | 3.0 | 117.0 | 3.0 |
| 12 | 4.0 | 1.0 | 31.0 | 2.0 | 0.0 | 0.0 | 0.0 | 3.0 | 26.0 | 9.0 | 396.0 | 18.0 | 2.0 | 0.0 | 6.0 | 0.0 |
| 13 | 5.0 | 23.0 | 2.0 | 2.0 | 1.0 | 6.0 | 2.0 | 1.0 | 40.0 | 329.0 | 34.0 | 30.0 | 2.0 | 14.0 | 6.0 | 1.0 |
| 14 | 8.0 | 13.0 | 25.0 | 2.0 | 67.0 | 143.0 | 74.0 | 88.0 | 5.0 | 25.0 | 6.0 | 8.0 | 8.0 | 14.0 | 10.0 | 2.0 |
| 15 | 10.0 | 7.0 | 53.0 | 18.0 | 30.0 | 29.0 | 84.0 | 52.0 | 12.0 | 12.0 | 73.0 | 18.0 | 6.0 | 5.0 | 66.0 | 23.0 |
| 16 | 7.0 | 4.0 | 40.0 | 7.0 | 14.0 | 9.0 | 20.0 | 10.0 | 44.0 | 37.0 | 173.0 | 22.0 | 8.0 | 24.0 | 73.0 | 6.0 |
| 17 | 13.0 | 8.0 | 38.0 | 14.0 | 9.0 | 14.0 | 27.0 | 24.0 | 66.0 | 74.0 | 139.0 | 27.0 | 7.0 | 7.0 | 29.0 | 2.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -0.486 | -0.784 | 0.489 | -1.451 | 0.144 | -0.07 | 0.248 | -0.934 | 0.715 | 0.469 | 1.149 | -0.539 | -1.596 | -1.211 | 0.171 | -1.451 |
| 02 | -0.036 | -1.11 | 0.804 | -0.717 | -0.216 | -0.342 | 0.32 | -0.934 | 0.171 | 0.343 | 1.397 | -0.486 | -1.766 | -1.11 | -0.342 | -1.451 |
| 03 | -0.435 | -0.388 | 0.296 | -0.784 | -0.256 | -0.298 | -1.211 | -0.036 | 0.699 | 1.054 | 1.108 | -0.784 | -1.596 | -0.654 | -0.14 | -1.971 |
| 04 | 0.223 | -3.122 | 0.715 | -1.211 | 0.449 | -1.324 | 0.546 | 0.223 | 0.65 | -1.018 | 0.087 | 0.775 | -0.784 | -3.122 | 0.248 | -1.451 |
| 05 | -1.971 | -4.397 | 1.615 | -4.397 | -3.122 | -4.397 | -0.435 | -4.397 | -2.58 | -2.58 | 1.783 | -3.122 | -2.58 | -3.122 | 1.34 | -4.397 |
| 06 | -4.397 | -4.397 | -1.211 | -4.397 | -4.397 | -4.397 | -2.23 | -4.397 | -3.122 | -4.397 | 2.726 | -2.23 | -4.397 | -4.397 | -3.122 | -4.397 |
| 07 | -4.397 | -4.397 | -3.122 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | -2.58 | -2.58 | 2.745 | -4.397 | -4.397 | -4.397 | -2.23 | -4.397 |
| 08 | -4.397 | -4.397 | -4.397 | -2.58 | -4.397 | -4.397 | -4.397 | -2.58 | 0.582 | 2.291 | -4.397 | 1.397 | -4.397 | -4.397 | -4.397 | -4.397 |
| 09 | -1.324 | -4.397 | 0.429 | -4.397 | -0.856 | -3.122 | 2.228 | -1.766 | -4.397 | -4.397 | -4.397 | -4.397 | -2.23 | -4.397 | 1.389 | -2.58 |
| 10 | -4.397 | -4.397 | -0.342 | -2.58 | -4.397 | -4.397 | -4.397 | -3.122 | -0.934 | -4.397 | 2.356 | 1.357 | -4.397 | -4.397 | -1.451 | -4.397 |
| 11 | -4.397 | -4.397 | -0.934 | -4.397 | -4.397 | -4.397 | -4.397 | -4.397 | 0.144 | -4.397 | 2.319 | -1.766 | -2.58 | -2.23 | 1.315 | -2.23 |
| 12 | -1.971 | -3.122 | -0.004 | -2.58 | -4.397 | -4.397 | -4.397 | -2.23 | -0.177 | -1.211 | 2.532 | -0.539 | -2.58 | -4.397 | -1.596 | -4.397 |
| 13 | -1.766 | -0.298 | -2.58 | -2.58 | -3.122 | -1.596 | -2.58 | -3.122 | 0.248 | 2.347 | 0.087 | -0.036 | -2.58 | -0.784 | -1.596 | -3.122 |
| 14 | -1.324 | -0.856 | -0.216 | -2.58 | 0.76 | 1.515 | 0.859 | 1.031 | -1.766 | -0.216 | -1.596 | -1.324 | -1.324 | -0.784 | -1.11 | -2.58 |
| 15 | -1.11 | -1.451 | 0.527 | -0.539 | -0.036 | -0.07 | 0.985 | 0.508 | -0.934 | -0.934 | 0.845 | -0.539 | -1.596 | -1.766 | 0.745 | -0.298 |
| 16 | -1.451 | -1.971 | 0.248 | -1.451 | -0.784 | -1.211 | -0.435 | -1.11 | 0.343 | 0.171 | 1.705 | -0.342 | -1.324 | -0.256 | 0.845 | -1.596 |
| 17 | -0.856 | -1.324 | 0.197 | -0.784 | -1.211 | -0.784 | -0.14 | -0.256 | 0.745 | 0.859 | 1.487 | -0.14 | -1.451 | -1.451 | -0.07 | -2.58 |