Model info
| Transcription factor | Znf143 | ||||||||
| Model | ZN143_MOUSE.H11DI.0.A | ||||||||
| Model type | Dinucleotide PWM | ||||||||
| LOGO | 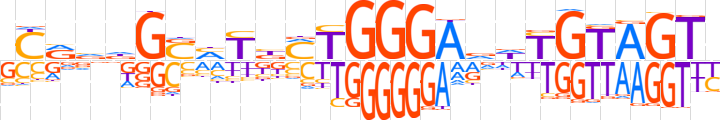 | ||||||||
| LOGO (reverse complement) | 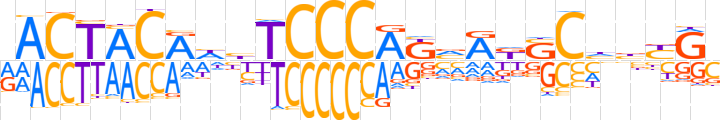 | ||||||||
| Data source | ChIP-Seq | ||||||||
| Model release | HOCOMOCOv11 | ||||||||
| Model length | 25 | ||||||||
| Quality | A | ||||||||
| Motif rank | 0 | ||||||||
| Consensus | SCRvdGSMYbMTGGGAvdYGTAGTY | ||||||||
| Best auROC (human) | 0.999 | ||||||||
| Best auROC (mouse) | 0.992 | ||||||||
| Peak sets in benchmark (human) | 28 | ||||||||
| Peak sets in benchmark (mouse) | 9 | ||||||||
| Aligned words | 465 | ||||||||
| TF family | More than 3 adjacent zinc finger factors {2.3.3} | ||||||||
| TF subfamily | ZNF76-like factors {2.3.3.28} | ||||||||
| MGI | MGI:1277969 | ||||||||
| EntrezGene | GeneID:20841 (SSTAR profile) | ||||||||
| UniProt ID | ZN143_MOUSE | ||||||||
| UniProt AC | O70230 (TFClass) | ||||||||
| Comment | |||||||||
| Downloads | pcm
pwm alignment threshold to P-value grid | ||||||||
| Standard thresholds |
| ||||||||
| GTEx tissue expression atlas | Znf143 expression | ||||||||
| Motifs in JASPAR |
PCM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | 0.0 | 17.0 | 2.0 | 1.0 | 7.0 | 82.0 | 4.0 | 16.0 | 18.0 | 270.0 | 8.0 | 18.0 | 1.0 | 14.0 | 1.0 | 4.0 |
| 02 | 12.0 | 1.0 | 13.0 | 0.0 | 116.0 | 24.0 | 216.0 | 27.0 | 7.0 | 2.0 | 5.0 | 1.0 | 7.0 | 5.0 | 27.0 | 0.0 |
| 03 | 43.0 | 26.0 | 64.0 | 9.0 | 12.0 | 2.0 | 10.0 | 8.0 | 50.0 | 55.0 | 132.0 | 24.0 | 7.0 | 5.0 | 11.0 | 5.0 |
| 04 | 20.0 | 5.0 | 55.0 | 32.0 | 21.0 | 2.0 | 36.0 | 29.0 | 61.0 | 14.0 | 86.0 | 56.0 | 3.0 | 1.0 | 21.0 | 21.0 |
| 05 | 2.0 | 0.0 | 102.0 | 1.0 | 0.0 | 0.0 | 22.0 | 0.0 | 5.0 | 4.0 | 181.0 | 8.0 | 0.0 | 0.0 | 135.0 | 3.0 |
| 06 | 0.0 | 4.0 | 3.0 | 0.0 | 0.0 | 0.0 | 4.0 | 0.0 | 28.0 | 321.0 | 66.0 | 25.0 | 0.0 | 8.0 | 3.0 | 1.0 |
| 07 | 20.0 | 7.0 | 0.0 | 1.0 | 192.0 | 95.0 | 14.0 | 32.0 | 17.0 | 47.0 | 4.0 | 8.0 | 7.0 | 10.0 | 8.0 | 1.0 |
| 08 | 1.0 | 63.0 | 7.0 | 165.0 | 3.0 | 35.0 | 1.0 | 120.0 | 1.0 | 9.0 | 0.0 | 16.0 | 0.0 | 8.0 | 1.0 | 33.0 |
| 09 | 2.0 | 0.0 | 3.0 | 0.0 | 5.0 | 21.0 | 48.0 | 41.0 | 1.0 | 2.0 | 5.0 | 1.0 | 4.0 | 67.0 | 137.0 | 126.0 |
| 10 | 2.0 | 7.0 | 1.0 | 2.0 | 30.0 | 51.0 | 3.0 | 6.0 | 18.0 | 164.0 | 5.0 | 6.0 | 7.0 | 117.0 | 38.0 | 6.0 |
| 11 | 1.0 | 23.0 | 0.0 | 33.0 | 3.0 | 62.0 | 2.0 | 272.0 | 0.0 | 3.0 | 0.0 | 44.0 | 0.0 | 6.0 | 1.0 | 13.0 |
| 12 | 0.0 | 0.0 | 4.0 | 0.0 | 0.0 | 0.0 | 94.0 | 0.0 | 0.0 | 0.0 | 3.0 | 0.0 | 2.0 | 0.0 | 360.0 | 0.0 |
| 13 | 0.0 | 0.0 | 2.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 459.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 |
| 14 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 0.0 | 0.0 | 4.0 | 1.0 | 454.0 | 2.0 | 0.0 | 0.0 | 1.0 | 0.0 |
| 15 | 4.0 | 0.0 | 0.0 | 0.0 | 1.0 | 0.0 | 0.0 | 0.0 | 411.0 | 7.0 | 14.0 | 24.0 | 0.0 | 2.0 | 0.0 | 0.0 |
| 16 | 176.0 | 49.0 | 156.0 | 35.0 | 5.0 | 2.0 | 0.0 | 2.0 | 5.0 | 3.0 | 5.0 | 1.0 | 14.0 | 2.0 | 4.0 | 4.0 |
| 17 | 102.0 | 11.0 | 35.0 | 52.0 | 5.0 | 3.0 | 4.0 | 44.0 | 23.0 | 21.0 | 10.0 | 111.0 | 4.0 | 0.0 | 3.0 | 35.0 |
| 18 | 0.0 | 20.0 | 6.0 | 108.0 | 3.0 | 5.0 | 4.0 | 23.0 | 4.0 | 20.0 | 3.0 | 25.0 | 4.0 | 13.0 | 16.0 | 209.0 |
| 19 | 2.0 | 0.0 | 9.0 | 0.0 | 5.0 | 1.0 | 51.0 | 1.0 | 3.0 | 3.0 | 23.0 | 0.0 | 5.0 | 1.0 | 358.0 | 1.0 |
| 20 | 0.0 | 5.0 | 6.0 | 4.0 | 1.0 | 1.0 | 3.0 | 0.0 | 13.0 | 10.0 | 31.0 | 387.0 | 0.0 | 1.0 | 1.0 | 0.0 |
| 21 | 11.0 | 1.0 | 2.0 | 0.0 | 9.0 | 4.0 | 3.0 | 1.0 | 20.0 | 8.0 | 12.0 | 1.0 | 367.0 | 0.0 | 22.0 | 2.0 |
| 22 | 0.0 | 3.0 | 402.0 | 2.0 | 3.0 | 5.0 | 3.0 | 2.0 | 4.0 | 3.0 | 30.0 | 2.0 | 0.0 | 1.0 | 3.0 | 0.0 |
| 23 | 1.0 | 0.0 | 5.0 | 1.0 | 1.0 | 3.0 | 4.0 | 4.0 | 1.0 | 5.0 | 11.0 | 421.0 | 2.0 | 1.0 | 1.0 | 2.0 |
| 24 | 3.0 | 0.0 | 0.0 | 2.0 | 1.0 | 1.0 | 2.0 | 5.0 | 3.0 | 9.0 | 6.0 | 3.0 | 7.0 | 194.0 | 18.0 | 209.0 |
PWM
| AA | AC | AG | AT | CA | CC | CG | CT | GA | GC | GG | GT | TA | TC | TG | TT | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 01 | -4.336 | -0.523 | -2.51 | -3.054 | -1.379 | 1.033 | -1.9 | -0.582 | -0.467 | 2.222 | -1.252 | -0.467 | -3.054 | -0.712 | -3.054 | -1.9 |
| 02 | -0.862 | -3.054 | -0.784 | -4.336 | 1.379 | -0.184 | 1.999 | -0.068 | -1.379 | -2.51 | -1.695 | -3.054 | -1.379 | -1.695 | -0.068 | -4.336 |
| 03 | 0.392 | -0.106 | 0.787 | -1.139 | -0.862 | -2.51 | -1.038 | -1.252 | 0.541 | 0.636 | 1.507 | -0.184 | -1.379 | -1.695 | -0.946 | -1.695 |
| 04 | -0.364 | -1.695 | 0.636 | 0.099 | -0.316 | -2.51 | 0.216 | 0.002 | 0.739 | -0.712 | 1.08 | 0.654 | -2.159 | -3.054 | -0.316 | -0.316 |
| 05 | -2.51 | -4.336 | 1.25 | -3.054 | -4.336 | -4.336 | -0.27 | -4.336 | -1.695 | -1.9 | 1.822 | -1.252 | -4.336 | -4.336 | 1.53 | -2.159 |
| 06 | -4.336 | -1.9 | -2.159 | -4.336 | -4.336 | -4.336 | -1.9 | -4.336 | -0.032 | 2.394 | 0.817 | -0.144 | -4.336 | -1.252 | -2.159 | -3.054 |
| 07 | -0.364 | -1.379 | -4.336 | -3.054 | 1.881 | 1.18 | -0.712 | 0.099 | -0.523 | 0.48 | -1.9 | -1.252 | -1.379 | -1.038 | -1.252 | -3.054 |
| 08 | -3.054 | 0.771 | -1.379 | 1.73 | -2.159 | 0.188 | -3.054 | 1.412 | -3.054 | -1.139 | -4.336 | -0.582 | -4.336 | -1.252 | -3.054 | 0.13 |
| 09 | -2.51 | -4.336 | -2.159 | -4.336 | -1.695 | -0.316 | 0.501 | 0.345 | -3.054 | -2.51 | -1.695 | -3.054 | -1.9 | 0.832 | 1.544 | 1.461 |
| 10 | -2.51 | -1.379 | -3.054 | -2.51 | 0.036 | 0.561 | -2.159 | -1.525 | -0.467 | 1.724 | -1.695 | -1.525 | -1.379 | 1.387 | 0.269 | -1.525 |
| 11 | -3.054 | -0.226 | -4.336 | 0.13 | -2.159 | 0.755 | -2.51 | 2.229 | -4.336 | -2.159 | -4.336 | 0.415 | -4.336 | -1.525 | -3.054 | -0.784 |
| 12 | -4.336 | -4.336 | -1.9 | -4.336 | -4.336 | -4.336 | 1.169 | -4.336 | -4.336 | -4.336 | -2.159 | -4.336 | -2.51 | -4.336 | 2.509 | -4.336 |
| 13 | -4.336 | -4.336 | -2.51 | -4.336 | -4.336 | -4.336 | -4.336 | -4.336 | -3.054 | -4.336 | 2.752 | -3.054 | -4.336 | -4.336 | -4.336 | -4.336 |
| 14 | -4.336 | -4.336 | -3.054 | -4.336 | -4.336 | -4.336 | -4.336 | -4.336 | -1.9 | -3.054 | 2.741 | -2.51 | -4.336 | -4.336 | -3.054 | -4.336 |
| 15 | -1.9 | -4.336 | -4.336 | -4.336 | -3.054 | -4.336 | -4.336 | -4.336 | 2.641 | -1.379 | -0.712 | -0.184 | -4.336 | -2.51 | -4.336 | -4.336 |
| 16 | 1.794 | 0.521 | 1.674 | 0.188 | -1.695 | -2.51 | -4.336 | -2.51 | -1.695 | -2.159 | -1.695 | -3.054 | -0.712 | -2.51 | -1.9 | -1.9 |
| 17 | 1.25 | -0.946 | 0.188 | 0.58 | -1.695 | -2.159 | -1.9 | 0.415 | -0.226 | -0.316 | -1.038 | 1.335 | -1.9 | -4.336 | -2.159 | 0.188 |
| 18 | -4.336 | -0.364 | -1.525 | 1.307 | -2.159 | -1.695 | -1.9 | -0.226 | -1.9 | -0.364 | -2.159 | -0.144 | -1.9 | -0.784 | -0.582 | 1.966 |
| 19 | -2.51 | -4.336 | -1.139 | -4.336 | -1.695 | -3.054 | 0.561 | -3.054 | -2.159 | -2.159 | -0.226 | -4.336 | -1.695 | -3.054 | 2.503 | -3.054 |
| 20 | -4.336 | -1.695 | -1.525 | -1.9 | -3.054 | -3.054 | -2.159 | -4.336 | -0.784 | -1.038 | 0.068 | 2.581 | -4.336 | -3.054 | -3.054 | -4.336 |
| 21 | -0.946 | -3.054 | -2.51 | -4.336 | -1.139 | -1.9 | -2.159 | -3.054 | -0.364 | -1.252 | -0.862 | -3.054 | 2.528 | -4.336 | -0.27 | -2.51 |
| 22 | -4.336 | -2.159 | 2.619 | -2.51 | -2.159 | -1.695 | -2.159 | -2.51 | -1.9 | -2.159 | 0.036 | -2.51 | -4.336 | -3.054 | -2.159 | -4.336 |
| 23 | -3.054 | -4.336 | -1.695 | -3.054 | -3.054 | -2.159 | -1.9 | -1.9 | -3.054 | -1.695 | -0.946 | 2.665 | -2.51 | -3.054 | -3.054 | -2.51 |
| 24 | -2.159 | -4.336 | -4.336 | -2.51 | -3.054 | -3.054 | -2.51 | -1.695 | -2.159 | -1.139 | -1.525 | -2.159 | -1.379 | 1.892 | -0.467 | 1.966 |